大規模データから新規抗菌物質を同定:腸内ウイルスのビッグデータを使った新しい治療法を開発
ー腸内ファージのデータベースを構築ー
大規模データから新規抗菌物質を同定:腸内ウイルスのビッグデータを使った新しい治療法を開発
―腸内ファージのデータベースを構築―
Cell Host & Microbe(2020年7月11日) DOI:10.1016/j.chom.2020.06.005
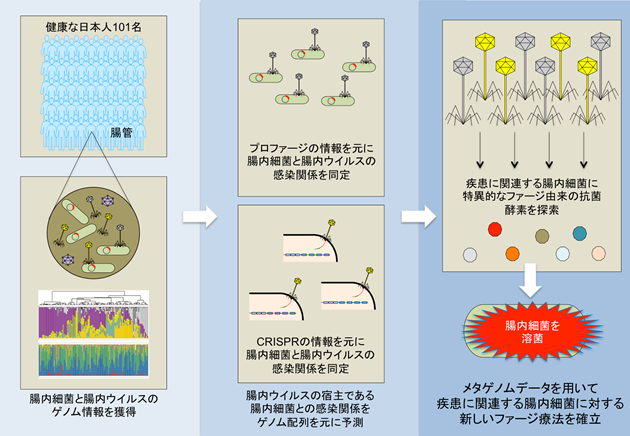
大阪市立大学大学院医学研究科 ゲノム免疫学の植松智教授(東京大学医科学研究所附属ヒトゲノム解析センター メタゲノム医学分野 特任教授、附属国際粘膜ワクチン開発研究センター 自然免疫制御分野 特任教授を兼務)、藤本康介助教(東京大学医科学研究所附属ヒトゲノム解析センター メタゲノム医学分野 特任助教、附属国際粘膜ワクチン開発研究センター 自然免疫制御分野 特任助教を兼務)、東京大学医科学研究所附属ヒトゲノム解析センター 健康医療インテリジェンス分野の井元清哉教授らの国際共同研究グループは、腸内細菌ゲノムと腸内ウイルスゲノムを網羅的に解析することで疾患特異的な腸内細菌の制御を可能とする新規抗菌物質の同定に成功しました。
近年では、腸内微生物ゲノム解析研究が進み、さまざまな疾患と腸内細菌叢の乱れとの関連性や、疾患の発症に直接的に関わる病原常在腸内細菌 (共生病原菌、Pathobiont)が次々と発見されています。一方、腸管内には腸内細菌だけでなく腸内ウイルスが大量に存在し、腸管の恒常性の維持に寄与していると考えられていますが、腸内ウイルスの解析は非常に難しく、これまで腸内ウイルス叢の詳細は明らかにされていませんでした。
本研究グループは、腸内ウイルスゲノムの解析パイプラインを独自に作成することで、これまでウイルス暗黒物質(viral dark matter※1)として解析が困難であったウイルスゲノムの詳細な分類が可能となり、日本人健常者の腸内ウイルス叢の全容が明らかとなりました。腸内細菌の大半を占める腸内ファージゲノムと腸内細菌ゲノムを組み合わせて解析することで、腸内ファージの宿主が特定でき、その情報を元に難病を引き起こす腸内常在細菌の一つであるClostridioides difficileに対するファージ由来の新しい抗菌物質を複数同定しました。
本研究成果は2020年7月11日(土)0時(日本時間)に国際科学雑誌『Cell Host & Microbe』 (IF=15.923)オンライン版に掲載されました。
【著者所属一覧】
1Department of Immunology and Genomics, Osaka City University Graduate School of Medicine, Osaka 545-8585, Japan
2Division of Metagenome Medicine, Human Genome Center, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
3Division of Innate Immune Regulation, International Research and Development Center for Mucosal Vaccines, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
4Division of Systems Immunology, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
5Mucosal Vaccine Project, BIKEN Innovative Vaccine Research Alliance Laboratories, Research Institute for Microbial Diseases, Osaka University, Osaka 565-0871, Japan
6Laboratory of DNA Information Analysis, Human Genome Center, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
7Division of Mucosal Immunology, Department of Microbiology and Immunology, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
8Division of Clinical Genome Research, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
9Department of Computer Science, Tokyo Institute of Technology, Tokyo 152-8550, Japan
10Department of Medicine, University of California San Diego, La Jolla, CA 92093, USA
11Division of Gastroenterology, Department of Medicine, CU-UCSD Center for Mucosal Immunology, Allergy and Vaccines, University of California San Diego, La Jolla, CA 92093, USA
12Division of Comparative Pathology and Medicine, Department of Pathology, University of California San Diego, La Jolla, CA 92093, USA
13Center for Veterinary Sciences and Comparative Medicine, University of California San Diego, La Jolla, CA 92093, USA
14Department of Mucosal Immunology, IMSUT Distinguished Professor Unit, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
15International Research and Development Center for Mucosal Vaccines, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
16Division of Health Medical Data Science, Health Intelligence Center, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
17Collaborative Research Institute for Innovative Microbiology, The University of Tokyo, Tokyo 113-8657, Japan
18Lead contact
