| 発表のポイント |
|---|
|
概要
大阪市立大学大学院医学研究科 ゲノム免疫学の植松智教授(東京大学医科学研究所附属ヒトゲノム解析センター メタゲノム医学分野 特任教授、附属国際粘膜ワクチン開発研究センター 自然免疫制御分野 特任教授を兼務)、藤本康介助教(東京大学医科学研究所附属ヒトゲノム解析センター メタゲノム医学分野 特任助教、附属国際粘膜ワクチン開発研究センター 自然免疫制御分野 特任助教を兼務)、東京大学医科学研究所附属ヒトゲノム解析センター 健康医療インテリジェンス分野の井元清哉教授らの国際共同研究グループは、腸内細菌ゲノムと腸内ウイルスゲノムを網羅的に解析することで疾患特異的な腸内細菌の制御を可能とする新規抗菌物質の同定に成功しました。近年では、腸内微生物ゲノム解析研究が進み、さまざまな疾患と腸内細菌叢の乱れとの関連性や、疾患の発症に直接的に関わる病原常在腸内細菌 (共生病原菌、Pathobiont)が次々と発見されています。一方、腸管内には腸内細菌だけでなく腸内ウイルスが大量に存在し、腸管の恒常性の維持に寄与していると考えられていますが、腸内ウイルスの解析は非常に難しく、これまで腸内ウイルス叢の詳細は明らかにされていませんでした。
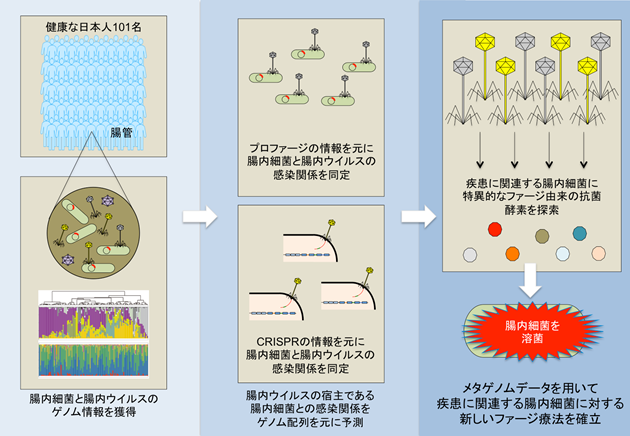
本研究グループは、腸内ウイルスゲノムの解析パイプラインを独自に作成することで、これまでウイルス暗黒物質(viral dark matter※1)として解析が困難であったウイルスゲノムの詳細な分類が可能となり、日本人健常者の腸内ウイルス叢の全容が明らかとなりました。腸内細菌の大半を占める腸内ファージゲノムと腸内細菌ゲノムを組み合わせて解析することで、腸内ファージの宿主が特定でき、その情報を元に難病を引き起こす腸内常在細菌の一つであるClostridioides difficileに対するファージ由来の新しい抗菌物質を複数同定しました。
本研究成果は2020年7月11日(土)0時(日本時間)に国際科学雑誌『Cell Host & Microbe』(IF=15.923)オンライン版に掲載されました。
※1 viral dark matter…腸管内に大量にあるはずのウイルスゲノムの解析が非常に困難であることを例えて、腸管内に暗黒物質があるとこれまで言われてきた(Elife. 2015 22;4:e08490., Cell Host Microbe. 2019 11;26:764-778.e5.)。
研究の背景
消化管内には細菌やウイルスをはじめとした常在微生物叢が存在し、私たちの健康に大きな影響を与えています。次世代シークエンサー※2をはじめとしたゲノム解析技術の進歩に伴い、常在微生物叢解析が盛んに行われるようになりました。特に、腸内細菌叢の乱れと疾患(肥満、糖尿病、関節リウマチ、炎症性腸疾患など)の関係性が明らかとなってきただけでなく、疾患の発症と直接的に関わるPathobiontも発見され、疾患の発症予防のために除菌が期待されています。しかしながら、抗生物質の使用は、有益菌も殺傷し、腸内細菌の乱れを助長する可能性があるため、Pathobiontだけを特異的に排除できる方法が求められています。また一方で、腸管内には細菌よりはるかに多い数のウイルスが存在しています。常在腸内ウイルスはインフルエンザウイルスのように私たちに感染するウイルスではなく、腸内細菌を宿主とするバクテリオファージ(ファージ)が主となります。したがって、腸内ウイルスを解析することでPathobiontに感染可能なファージが同定され、その情報を基盤として新しいPathobiontの制御法が見出される可能性があります。
しかし、腸内ウイルスの網羅的な解析をするためには、全ゲノムシークエンスが必要であること、これまで腸内ウイルスの大規模なデータベースが存在していないためゲノム配列を取得してもレファレンスが非常に乏しく解析が難しいこと、さらにファージ学が元々ファージの形態を基盤として発展してきた学問であるためゲノムによるファージ分類法がきちんと確立されていないことなどから、腸内ウイルス叢の全容はこれまでほとんど明らかとなっていませんでした。
※2 次世代シークエンサー…遺伝子の配列を高速に読み出せる装置で、圧倒的に短時間かつ低コストでゲノム解析をすることが可能。
研究の内容
本研究では、101名の日本人健常者の糞便サンプルから腸内細菌ゲノムと腸内ウイルスゲノムを抽出し、それぞれ全ゲノム解析を行いました。これまで腸内ウイルス叢のゲノム解析手法が確立されていなかったため、東京大学医科学研究所のスーパーコンピュータ上でゲノムアセンブリを行い、擬似クローニングをすることで確度の高い腸内ウイルス解析パイプラインを構築しました。その解析パイプラインを用いて健常者の腸内ウイルス叢を解析したところ、健常者の腸内ウイルス叢は個々人で非常にユニークな構成をしていることが明らかとなりました(図1)。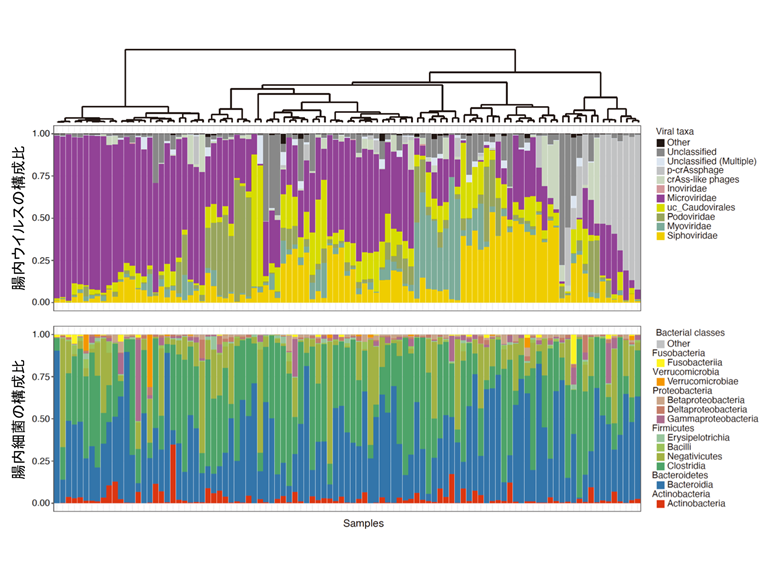
図1:101名の腸内ウイルス叢(上段)と細菌叢(下段)の構成比
腸内ウイルスの主となるファージは溶菌性ファージと溶原性ファージの2種類に大別され、それぞれ異なる生活環を呈します。溶菌性ファージは宿主細菌に吸着して感染した後、自身のゲノムを細菌に注入・複製し、溶菌することで娘ファージを菌体外に放出します。この時、細菌ゲノム内にCRISPR配列が挿入されることで、感染履歴が記憶されます(細菌の防御応答)。
一方、溶原性ファージは宿主細菌に感染し自身のゲノムを注入した後、宿主細菌のゲノムに自らのゲノムを組み込むことができます。この状態はプロファージと呼ばれ、宿主細菌と共に増殖することが可能となるだけでなく、宿主細菌が老化した場合などは溶菌性ファージと同様に菌体外へと放出されます。
本研究では、得られたゲノム情報を利用して、腸内細菌と腸内ウイルスのプロファージ配列に着目した感染関係とCRISPR配列に着目した感染関係を同定する解析パイプラインを構築しました。
さらに今回得られた健常者のシークエンスデータおよびこれまで臨床分離株として単離されているClostridioides difficile(偽膜性腸炎の起因菌として知られている菌)の17菌株のシークエンスデータを用いて、C. difficile特異的なプロファージ配列を抽出しました。
そのプロファージ配列から、ファージが菌体内で増殖した後に菌体外に放出される際に使用される溶菌酵素として知られるエンドライシンの配列を探索し、新規配列を複数同定しました。それらのエンドライシンを合成し、in vitroで溶菌活性を有することを示しただけでなく、C. difficile感染マウスモデルにおいて今回新しく同定したエンドライシンが効果を示すことを明らかとしました(図2)。本研究により、健常者の同一糞便における腸内ウイルス叢と細菌叢のメタゲノムデータベースを世界に先駆けて作成することができました。
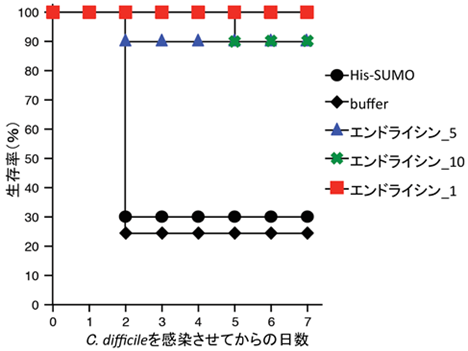
図2:新しく同定したエンドライシンは致死性C. difficile感染症マウスモデルの生存率を著明に改善する
期待される効果
これまで解析が困難であった腸内細菌と腸内ファージの感染関係を明らかにすることで、C. difficileに対する新しい溶菌酵素を同定することができました。この手法を疾患特異的な腸内細菌を標的として応用することで、新しい溶菌酵素の同定につながり、これまで制御できなかった腸内細菌叢の乱れに関連するさまざまな難治性の疾患に対する新たな治療アプローチとして使える可能性が期待されます。資金・特許等について
本研究は、武田科学振興財団研究助成、キヤノン財団研究助成、JST COI STREAM東京大学V1拠点「自分で守る健康社会」、HPCI戦略プログラム 分野1「予測する生命科学・医療および創薬基盤」の一環として行われました。東京大学医科学研究所システム免疫学社会連携研究部門に対して、株式会社医学生物学研究所の支援を受けました。発表雑誌
雑誌名:「Cell Host & Microbe」 7月11日オンライン版(IF=15.923)
論文タイトル:Metagenome Data on Intestinal Phage–Bacteria Associations Aids the Development of Phage Therapy Against Pathobionts
DOI:10.1016/j.chom.2020.06.005
著者:Kosuke Fujimoto1,2,3, Yasumasa Kimura4, Masaki Shimohigoshi1, Takeshi Satoh4, Shintaro Sato1,5, Georg Tremmel6, Miho Uematsu1, Yunosuke Kawaguchi1, Yuki Usui4, Yoshiko Nakano1, Tetsuya Hayashi1, Koji Kashima7, Yoshikazu Yuki7, Kiyoshi Yamaguchi8, Yoichi Furukawa8, Masanori Kakuta6, Yutaka Akiyama9, Rui Yamaguchi6, Sheila E. Crowe10, Peter B. Ernst11,12,13, Satoru Miyano6, Hiroshi Kiyono11,12,14,15, Seiya Imoto16,17*, Satoshi Uematsu1,2,3,17,18* (*責任著者)
【著者所属一覧】
1Department of Immunology and Genomics, Osaka City University Graduate School of Medicine, Osaka 545-8585, Japan
2Division of Metagenome Medicine, Human Genome Center, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
3Division of Innate Immune Regulation, International Research and Development Center for Mucosal Vaccines, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
4Division of Systems Immunology, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
5Mucosal Vaccine Project, BIKEN Innovative Vaccine Research Alliance Laboratories, Research Institute for Microbial Diseases, Osaka University, Osaka 565-0871, Japan
6Laboratory of DNA Information Analysis, Human Genome Center, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
7Division of Mucosal Immunology, Department of Microbiology and Immunology, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
8Division of Clinical Genome Research, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
9Department of Computer Science, Tokyo Institute of Technology, Tokyo 152-8550, Japan
10Department of Medicine, University of California San Diego, La Jolla, CA 92093, USA
11Division of Gastroenterology, Department of Medicine, CU-UCSD Center for Mucosal Immunology, Allergy and Vaccines, University of California San Diego, La Jolla, CA 92093, USA
12Division of Comparative Pathology and Medicine, Department of Pathology, University of California San Diego, La Jolla, CA 92093, USA
13Center for Veterinary Sciences and Comparative Medicine, University of California San Diego, La Jolla, CA 92093, USA
14Department of Mucosal Immunology, IMSUT Distinguished Professor Unit, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
15International Research and Development Center for Mucosal Vaccines, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
16Division of Health Medical Data Science, Health Intelligence Center, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
17Collaborative Research Institute for Innovative Microbiology, The University of Tokyo, Tokyo 113-8657, Japan
18Lead contact
【著者所属一覧】
1Department of Immunology and Genomics, Osaka City University Graduate School of Medicine, Osaka 545-8585, Japan
2Division of Metagenome Medicine, Human Genome Center, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
3Division of Innate Immune Regulation, International Research and Development Center for Mucosal Vaccines, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
4Division of Systems Immunology, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
5Mucosal Vaccine Project, BIKEN Innovative Vaccine Research Alliance Laboratories, Research Institute for Microbial Diseases, Osaka University, Osaka 565-0871, Japan
6Laboratory of DNA Information Analysis, Human Genome Center, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
7Division of Mucosal Immunology, Department of Microbiology and Immunology, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
8Division of Clinical Genome Research, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
9Department of Computer Science, Tokyo Institute of Technology, Tokyo 152-8550, Japan
10Department of Medicine, University of California San Diego, La Jolla, CA 92093, USA
11Division of Gastroenterology, Department of Medicine, CU-UCSD Center for Mucosal Immunology, Allergy and Vaccines, University of California San Diego, La Jolla, CA 92093, USA
12Division of Comparative Pathology and Medicine, Department of Pathology, University of California San Diego, La Jolla, CA 92093, USA
13Center for Veterinary Sciences and Comparative Medicine, University of California San Diego, La Jolla, CA 92093, USA
14Department of Mucosal Immunology, IMSUT Distinguished Professor Unit, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
15International Research and Development Center for Mucosal Vaccines, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
16Division of Health Medical Data Science, Health Intelligence Center, The Institute of Medical Science, The University of Tokyo, Tokyo 108-8639, Japan
17Collaborative Research Institute for Innovative Microbiology, The University of Tokyo, Tokyo 113-8657, Japan
18Lead contact
