| 発表のポイント |
|---|
|
概要
東北大学東北メディカル・メガバンク機構(ToMMo)※1、岩手医科大学いわて東北メディカル・メガバンク機構(IMM)※2、理化学研究所および東京大学医科学研究所は共同研究を実施し、計7,609人分のバリアントを含むバリアント頻度情報について、非制限公開データ※3の GEM Japan Whole Genome Aggregation(GEM-J WGA)パネルとして、科学技術振興機構(JST)バイオサイエンスデータベースセンター(NBDC)のTogoVar※4より2020年7月27日から公開します(図)。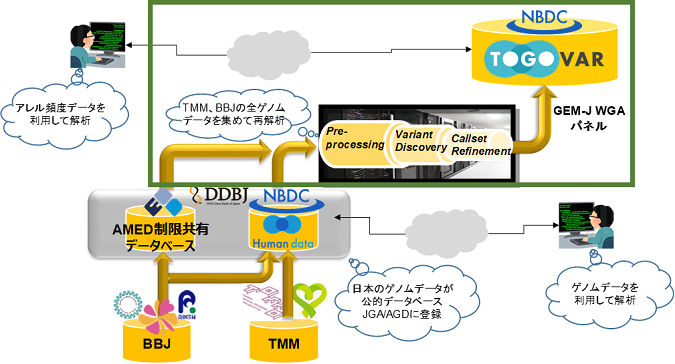
図.TMM、BBJの全ゲノムデータを再解析してGEM-J WGAパネルを作成するまでの流れ
これらのデータは、ToMMoおよびIMMの持つ4,495人分の全ゲノム配列(Whole Genome Sequence: WGS)※5情報と理化学研究所および東京大学医科学研究所が持つバイオバンク・ジャパン(BBJ)※6の3,114人分のWGS情報を合わせた計7,609人分のWGS情報を用いてバリアント※7検知を実施して得られたものです。また、解析する際に得られた個人ごとのゲノム配列を参照ゲノム配列※8にマッピングした結果およびバリアント情報は、制限公開/制限共有データ※3として、国立遺伝学研究所生命情報・DDBJセンターのJGA/AGD※9より近日中に公開する予定です。
背景
現在、次世代シークエンサー※10を用いたゲノム配列取得(シークエンス)解読コストの大幅低下に伴い、患者の検体(血液・組織等)から採取したDNAより得られたシークエンスデータから疾患に関与するバリアントを探索する等、疾患の特定や病態解明を目指すゲノム医療研究が進展しています。また、その成果の積み重ねにより、がん領域では一般診療レベルでのゲノム医療が実現しつつあります※11。ゲノム医療研究の研究協力者から取得した検体のWGS情報からは数百万個ものバリアントが検知されますが、これらのバリアントには疾患の原因となるバリアントと疾患の発症における意義が不明なバリアント(VUS: Variant of Uncertain Significance)とが混在しています。
単一遺伝子疾患の解析においては、一般集団が必要となります。そのため、WGSにより得られたバリアントのアレル頻度※12データベースを整備することが望ましいと考えられます。また、生物集団の遺伝的多様性を反映するバリアントのアレル頻度を変化させる要因は突然変異、遺伝的浮動※13、移住、自然選択とされていますが、バリアントの中でもアレル頻度の極めて低い(レアな)バリアントは集団内で浮動する(アレル頻度が変動する)※14ため、頻度フィルタ※15には、研究協力者と同じ遺伝的背景を持つ集団からのアレル頻度データを用いることが望ましいと考えられます。さらに、遺伝的浮動と移住は地域の影響を大きく受けるため、単一の地域のみならず日本各地域から取得し、より一般集団を反映した情報を得ることは日本のゲノム医療の実現に向けて極めて重要となります。
また、アレル頻度情報は、疾患の原因となるバリアント同定について研究する際に、他の集団のアレル頻度と比較することにも利用されますが、バリアント検知には様々のツールが開発、利用されているため、国際的な標準手法を採用し比較可能なデータを作成することも重要です。
さらに、WGSは全エクソーム※16と比較してゲノムの翻訳領域においてバイアスの少ない結果をもたらすと報告されており※17、医療応用に向けて多くの研究者がデータを利用できるように、WGSをもとにしたバリアント頻度情報のデータベースの整備が、イントロン領域※16も含めた網羅的な遺伝子異常を検出することに役立つとされています。
今回の成果
WGSをもとにしたバリアント頻度情報の解析には、東北メディカル・メガバンク計画による宮城県と岩手県の一般住民を対象としたコホート調査への協力者 4,307人分のデータに加えて、生活習慣病患者群の検体を収集するために理化学研究所および東京大学医科学研究所によって実施されたオーダーメイド医療実現化プロジェクトおよびオーダーメイド医療の実現プログラムの両事業(バイオバンク・ジャパン)※18に参加協力する病院から集められた患者(協力者)2,857人分、国立病院機構長崎医療センターにおける協力者188人、理化学研究所 生命医科学研究センターにおける協力者257人分のWGSデータが用いられました(表1)。
これらのWGSデータから国際的に比較可能なデータを作成するため、ToMMo内のスーパーコンピュータを用いて、GATK Best Practicesに準拠した方法※19により、GRCh37※8の参照ゲノム配列へのマッピングおよびバリアント検知を実施しました。
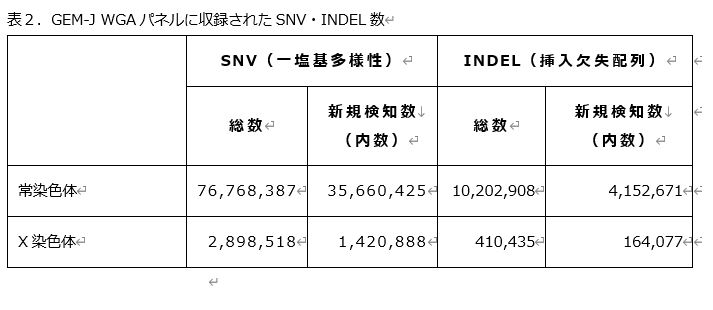
その結果、常染色体で76,768,387個の一塩基多様性(Single Nucleotide Variation: SNV)※20、10,202,908個の挿入欠失配列(InsertionおよびDeletion:INDEL)※21が検知されました。また、X染色体では2,898,518個のSNV、410,435個のINDELが検知されました (表2)。
また、得られた個人ごとのバリアント情報を元に国際1,000人ゲノムプロジェクト※22を参照した主成分分析を実施し、遺伝的背景の確認を行いました。さらに、個人ごとの遺伝的距離を比較することでバリアント頻度情報のバイアスとなる※23近親者の排除といった品質管理を実施しました。
このプロジェクトは、関係機関の賛同・協力を得てAMEDが提案したプロジェクトであり、ゲノム情報や臨床情報の国際的なデータシェアリングを推進しているThe Global Alliance for Genomics and Health(GA4GH)※24の基幹プロジェクトである GEnome Medical alliance Japan(GEM Japan, GEM-J)※25の取り組みの一つです。日本人集団のバリアント頻度パネルを公開することにより、難病・稀少疾患解明への国際的な貢献に資することが期待されます。
バリアント情報の解析は、国際1,000人ゲノムプロジェクトに始まり、gnomAD※26等、これまで多種多様な集団・集合体の解析がなされていますが、特定の民族集団である日本において、精度の高いゲノム診断を行い、ゲノム医療を展開するためには、数万人規模以上のデータが必要となります。
一方、グローバルなゲノム医療研究において、ヨーロッパ系のゲノムデータは多数公開されていますが、東アジア系のデータは少ない状況です。東アジア人のゲノム診断を行うために、あるいは多民族でのゲノム診断の「フィルタ」を行うために、日本人を含む東アジア人のデータ共有が求められています。
今後の展望
本プロジェクトで得られた結果は将来のゲノム医療に向けての基礎データになると期待されるほか、以下の研究に役立つことが期待されます。(1)頻度フィルタの精度向上による、難病・稀少疾患の原因バリアント同定精度の向上
(2)より人数が多い他の参照パネルとの比較によるレアハプロタイプ情報の取得、その取得によりレアバリアントのインピュテーション※27精度の向上※28。
今回の成果を踏まえ、精度の高いゲノム診断やゲノム医療の進展に資することを目指し、国内で解析の進むWGSデータを集め、10万人規模のバリアント頻度パネルへの拡大を検討しています。
用語解説
※1 東北大学東北メディカル・メガバンク機構(ToMMo)東日本大震災からの復興支援事業である東北メディカル・メガバンク計画(TMM)の一環として、AMEDの支援の下、岩手医科大学いわて東北メディカル・メガバンク機構とともに、岩手県・宮城県の被災地を中心にした大規模健康調査とゲノムコホート研究を行い、地域医療の復興に貢献するとともに、個別化医療・個別化予防などの次世代医療体制の構築を目指している。↑
(URL: https://www.megabank.tohoku.ac.jp/)
※2 岩手医科大学いわて東北メディカル・メガバンク機構(IMM)
東日本大震災からの復興支援事業であるTMMの一環として、AMEDの支援の下、ToMMoとともに、岩手県・宮城県の被災地を中心にした大規模健康調査とゲノムコホート研究を行い、地域医療の復興に貢献するとともに、個別化医療・個別化予防などの次世代医療体制の構築を目指している。↑
(URL: http://iwate-megabank.org/)
※3 非制限公開データ、制限公開データ、制限共有データ
非制限公開データとは、アクセスに制限を設けることなく、利用することが可能な公開データ。例えば、すでに発表された論文の集計・統計解析データ等が含まれる。
制限公開データとは、データ利用者、利用目的等を明らかにした上で、関連研究に従事したことのある研究者が研究のために利用することが可能な公開データ。
制限共有データとは、原則、データを所有する研究者と利用を希望する研究者間の合意に基づき利用可能な非公開データ。↑
「ゲノム医療実現のためのデータシェアリングポリシー」(https://www.amed.go.jp/content/000060867.pdf)を参照。
※4 TogoVar(日本人ゲノム多様性統合データベース)
主として日本人のバリアントのアレル頻度※13やバリアント※7と関連する疾患や文献の情報を収集・整理し、それらの情報をワンストップで取得可能なデータベース。JST ライフサイエンスデータベース統合推進事業の一環として、NBDCと情報・システム研究機構ライフサイエンス統合データベースセンター(DBCLS)が共同で開発。↑
(URL:https://togovar.biosciencedbc.jp)
※5 全ゲノム配列(Whole Genome Sequence: WGS)
ヒトの遺伝情報は、A、C、T、Gの4種類の塩基からなるDNA配列に保存されている。ヒトの遺伝情報全体をヒトゲノムというが、全ゲノム配列は遺伝情報全体を構成するDNA配列を指す。↑
※6 バイオバンク・ジャパン(BBJ)
アジア最大規模の疾患バイオバンクで、東京大学医科学研究所内に設置されている。オーダーメイド医療の実現プログラム・オーダメイド医療実現化プロジェクトにおいて約26.7万人の生活習慣病の患者からDNAや血清、臨床情報を収集・保管し、研究者へ試料やデータの提供を行っている。↑
(URL: https://biobankjp.org/)
※7 バリアント
ヒトゲノムはそのほとんど(99%以上)がすべてのヒトで同じだが、ごく一部だけ違いがある場所がある。この個人間の違いがある部分をバリアントという。↑
※8 参照ゲノム配列
国際的な学術組織The Genome Reference Consortiumが継続的に改訂を行っているヒトゲノムの塩基配列を指す。国際ヒトゲノム参照配列(あるいは基準ゲノム配列)ともいわれる。GRCh37(Genome Reference Consortium Human Build 37)は、2009年に発表され、次世代シークエンサーやマイクロアレイ等のゲノム解析に参照ゲノム配列として広く用いられている。↑
※9 JGA/AGD
Japanese Genotype-phenotype Archive (JGA) / AMED Genome group sharing Database (AGD) は、個人レベルの遺伝学的なデータと匿名化された表現型情報等を保存し、データの共有を可能にするデータベース。↑
(URL:https://www.ddbj.nig.ac.jp/jga/index.html, URL:https://www.ddbj.nig.ac.jp/agd/index.html)
制限公開/制限共有データ※3は、科学的観点と研究体制の妥当性に関する審査を経た上で、データの利用を承認された研究者に利用される。制限共有の場合は、事前にデータ提供者の許可を受ける必要がある(共同研究である必要はない)。JGAは制限公開のためのデータベースであり、AGDは制限共有のためのデータベース。データ提供・利用の申請はNBDCヒトデータベース(https://humandbs.biosciencedbc.jp/)。↑
※10 次世代シークエンサー
2000年半ばごろに登場した、DNAの塩基配列を同時並行に決定することができる機械のこと。従来の塩基配列決定法であったサンガーシークエンスでは解読配列の選別が必要だったが、次世代シークエンサーにおいては、選別を必要とせず、DNAサンプル中に存在する全ての断片の塩基配列を同時に並行して決定することができ、高速に、かつ大量にDNAの塩基配列を決定できるようになった。↑
※11がん領域での一般診療レベルのゲノム医療
がんは、ゲノムの変化に伴って塩基配列の違いなどが生じ、遺伝子が正常に機能しなくなった結果、起こる病気。がんに関係する多数の遺伝子を同時に調べる検査の一部が保険診療として2019年6月より実施可能となった。詳しくは、例えば https://ganjoho.jp/public/dia_tre/treatment/genomic_medicine/genmed02.html を参照。↑
※12アレル頻度
一つの遺伝子座に対して複数の対立遺伝子(アレル、allele)が存在する場合、それぞれの対立遺伝子の集団中における頻度。↑
※13 遺伝的浮動
ある集団内でのアレル頻度の変化をいう。集団中の遺伝的多様性を減少させる効果があり、集団が小さいときに強く働く。例えばヒトの血液型の違いはそれぞれ生存にとって有利不利がないので,現在民族間、国家間で血液型 (A、B、O、AB) の割合に違いがあるのは,偶然の変動、つまり遺伝的浮動の結果と考えられる。(ブリタニカ国際大百科事典 小項目事典より一部引用)↑
※14 アレル頻度の変動
偽陽性の原因となる進化論的中立なバリアントアレルにおいて、低頻度アレルは最近発生した突然変異によるものであることを意味し、アフリカ人、ヨーロッパ人、アジア人に分岐後に発生したものを多く含むと考えられるため、欧米系集団を主体とするExAC / gnomADデータベース(https://gnomad.broadinstitute.org/)には存在しないバリアントも多いと考えられる。↑
※15 頻度フィルタ
頻度フィルタを適切に行うことで、本来必要のないバリアントに対する介入により生じる無駄なコストや健康被害を防止することにつながる。↑
※16 全エクソーム
ヒトゲノムのうちタンパク質をコードするエクソン領域(エクソーム)全ての塩基配列。ちなみに、タンパク質(アミノ酸)に翻訳されない領域がイントロン領域。↑
※17 WGSは全エクソームと比較してゲノムの翻訳領域においてバイアスの少ない結果をもたらす
2015年Lelieveld らによる発表。DOI: 10.1002/humu.22813
https://www.ncbi.nlm.nih.gov/pubmed/25973577 ↑
※18 オーダーメイド医療実現化プロジェクト、オーダーメイド医療の実現プログラム
各事業の詳細については、 https://biobankjp.org/backnumber.html を参照。↑
※19 GATK Best Practicesに準拠した方法
GATK(Genome Analysis Toolkit)は 、米国ブロード研究所 (Broad Institute)で開発され、次世代シークエンサーから出力される大量の塩基配列データから遺伝子の変異解析を行う機能を備えた多数のプログラムから構成されている解析ツール群。国際1,000人ゲノムプロジェクト※22を始めゲノム解析に関する共同研究では、GATKを標準的な解析ツールと認知し、利用している。↑
(URL: https://github.com/gpc-gr/panel3552-scripts)
※20 一塩基多様性(Single Nucleotide Variation: SNV)
ゲノムの個人間の違いのうち、A、C、T、Gからなるヒトゲノム塩基配列上の1カ所の違い(置換)をいう。ある集団内での頻度が1%以上あるものを、別にSNP(Single Nucleotide Polymorphism)と呼ぶ。↑
※21 挿入欠失配列(InsertionおよびDeletion:INDEL)
挿入配列はゲノム配列の特定の位置に挿入された別の配列をいう。欠失配列はゲノム配列の一部が失われた配列をいう。↑
※22 国際1,000人ゲノムプロジェクト
人類集団の詳細な遺伝的多様性を行うことを目指し、世界各地の約1,000人の全ゲノムシークエンスを行った国際研究計画。当計画は、現在解析人数を2,535人にまで拡張したphase3が完了済み。詳細については、http://www.1000genomes.org を参照。↑
※23 バリアント頻度情報のバイアス
近親者はその他の人と比較し多くのバリアントを共有するため、数多くの近親者が含まれる場合はその近親者の集団に特異的に観察されるバリアントのアレル頻度が高くなり、一般集団のアレル頻度と異なるバイアスとなる。↑
※24 The Global Alliance for Genomics and Health (GA4GH)
研究参加者の同意や個人情報の保護等の配慮の下でゲノム情報等のデータシェアリングを可能とするための基盤的な枠組みの構築や技術的な国際標準の設定をすることを目的として、2013年に発足した国際協力組織(任意団体)。2019年時点で50カ国、500超の機関(日本からはAMEDを含む15機関)が加盟。↑
(URL: https://www.ga4gh.org/)
※25 GEnome Medical alliance Japan (GEM Japan, GEM-J)
データシェアリングを進めながらゲノム医療の実現を目指すAMEDの各事業に関わる大学、研究所、病院等と日本全国規模で協力体制を築き、臨床情報と個人ゲノム情報のデータシェアリングと研究利用を促進し、ゲノム医療の実現を目指す。具体的には、AMEDが策定した「ゲノム医療実現のためのデータシェアリングポリシー」※3の対象事業である、臨床ゲノム情報統合データベース整備事業(https://www.amed.go.jp/program/list/14/01/006.html)、ゲノム医療実現推進プラットフォーム事業(https://www.amed.go.jp/program/list/14/01/001.html)、ゲノム研究バイオバンク事業(https://www.amed.go.jp/program/list/14/01/003.html)やTMM※1にて取り組むこととしており、日本人全ゲノム解析に基づく日本人アレル頻度情報の公開、日本人の疾患関連バリアント情報の公開、GEM Japanワークショップの開催等による協力を進めている。
(URL: https://www.amed.go.jp/aboutus/collaboration/ga4gh_gem_japan.html)↑
※26 gnomAD(The Genome Aggregation Database)
米国ブロード研究所が提供する、様々な研究で調べられたヒトのエクソームやゲノムのデータを集約したデータベース。疾病研究で発見されたバリアントが一般的なバリアントなのか病気に特有のものなのかを調べるためのレファレンスなどとして活用されている。
(URL: https://gnomad.broadinstitute.org/)↑
※27 インピュテーション
DNAマイクロアレイで測定して得られた遺伝型を用いて、実験的に測定していない遺伝的変異をコンピュータで推定し、補完する遺伝統計学的手法。↑
※28インピュテーション精度の向上
多因子疾患に関連するレアバリアントは、進化論的に一掃されつつあるバリアントであることを意味すると思われ、一般にリスク効果が高いため、多因子疾患にかかわるレアバリアントの検出精度が高まることは、これについてのゲノム医療実現を加速するものと考えられる。
参考文献: McCarthy et al.: A reference panel of 64,976 haplotypes for genotype imputation, Nat Genet 2016, 48(10):1279-83. ↑
