| 発表のポイント |
|---|
|
概要
東京大学医科学研究所システムウイルス学分野の伊東潤平准教授および佐藤佳教授の研究チームは、SARS-CoV-2の適応度(流行拡大能力)(注1)をスパイクタンパク質配列から予測するAIモデル「CoVFit」を開発しました。CoVFitは、未知の変異株に対しても高い精度で適応度を予測できることから、ゲノム配列情報のみに基づいて、流行拡大のリスクが高い変異株を従来よりも早期に検出できると期待されます。本研究ではさらに、CoVFitを活用して、SARS-CoV-2の適応度上昇に寄与する変異を網羅的に同定し、ウイルスの進化に関わるメカニズムの一端を明らかにしました。最後に、CoVFitを用いるとSARS-CoV-2の単一アミノ酸置換による進化を比較的高い精度で予測できることを示しました。本研究成果は2025年5月13日、米国科学雑誌「Nature Communications」オンライン版で公開されました。
発表内容
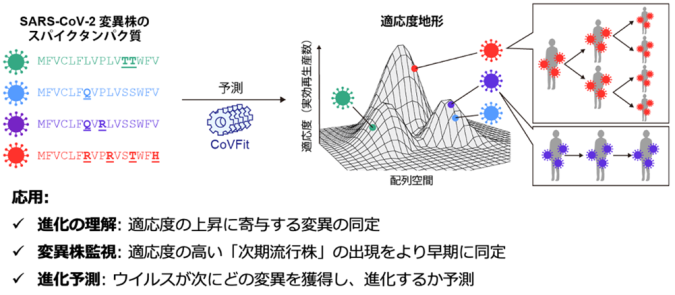
ウイルス感染症の制御が困難である主な要因の一つは、ウイルスが変異を通じて進化し、性質を変化させる点にあります。COVID-19パンデミックでは、「変異株」と呼ばれる多様な性質を持つSARS-CoV-2が次々と出現したことで、流行の制御が困難となりました。SARS-CoV-2の研究を通じて、ウイルスの進化と流行の原理を理解することができれば、COVID-19のみならず将来のパンデミックを含む多くの感染症対策に役立つ知見が得られると期待されます。SARS-CoV-2の流行動態の特徴として、適応度の高い変異株が出現すると、それが次期流行株となり、新たな感染の波を引き起こすというパターンが知られています。したがって、次期流行株の出現を流行の超初期段階で予測できれば、感染拡大を未然に防ぐ手がかりとなります。さらに、これらの株について免疫逃避能や病原性、治療薬耐性といった性質を早期に明らかにできれば、流行前の段階でそのリスクを評価することが可能となります。
SARS-CoV-2変異株の適応度は、その変異株の持つ変異パターンに依存して決定されるため、理論的には遺伝子配列から予測することが可能です。適応度予測モデルを確立することで、ゲノム配列が解読された段階で、流行を急速に拡大する可能性の高い変異株を早期に同定できると期待されます。また、SARS-CoV-2がこれまでに何度も流行を繰り返してきた原因の一つは、進化の過程でSARS-CoV-2の適応度が段階的に上昇してきたことにあります。適応度予測モデルを用いることで、SARS-CoV-2がどのような変異を獲得することで適応度を高めてきたのかを推定し、進化のメカニズムを明らかにすることが可能となります。さらに、SARS-CoV-2は自然選択の圧力の下で、適応度を高める方向に進化する傾向があるため、精度の高い適応度予測モデルが実現すれば、将来のウイルス進化の方向性を予測することも可能になると考えられます。
本研究では、スパイクタンパク質配列のみに基づいて変異株の適応度を予測するAIモデル「CoVFit」を開発しました。CoVFitは、大規模言語モデルの一種であるタンパク質言語モデル(注2)を基盤とし、i) ウイルスゲノム疫学データ(注3)に基づく変異株の適応度情報とスパイクタンパク質配列、ii) 実験により網羅的に測定された免疫逃避能に関する機能データを用いて訓練されたマルチタスク学習モデルです。このマルチタスク学習スキームにより、CoVFitは変異の機能情報を適応度予測に効果的に活用することができ、その結果、高精度な予測を実現しました。
本研究ではまず、未知の変異株に対する予測精度を検証するため、ウイルスゲノム疫学データをある時点に基づき分割し、過去のデータと未来のデータを作成しました。そして、過去データだけを用いてCoVFitを訓練し、過去のデータだけを学習したCoVFitを用いて未来の変異株の適応度を予測しました。その結果、CoVFitは未知の、未来の変異株の適応度を高い精度で予測できることが確認されました(図1)。
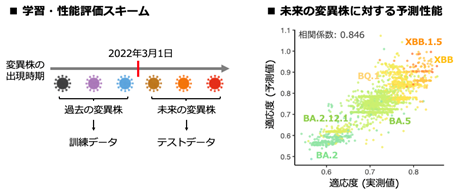
左) 変異株出現時期に基づくデータセットの分割と学習スキーム。右)未来の変異株に対する適応度の予測結果。
さらに本研究では、SARS-CoV-2の進化の過程で生じた適応度の上昇イベントを網羅的に解析し、進化の過程で959回の適応度上昇が発生していたことを明らかにしました。これらの適応度上昇に関与する変異は、主にスパイクタンパク質の受容体結合ドメイン(注4)に集中していました。この結果は、これらの変異が受容体結合能や中和抗体からの逃避能の向上により適応度の上昇に寄与することを示唆しています(図2)。
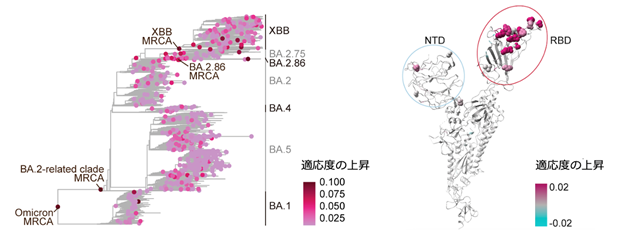
左)SARS-CoV-2進化過程における適応度上昇イベントの網羅的検出。右)適応度上昇に寄与する変異。
また本研究チームは、CoVFitを用いてウイルスの進化を予測する手法も開発しました。具体的には、ある変異株を基準に、単一アミノ酸置換を全パターン挿入した仮想変異体を網羅的に生成し、それらの適応度を予測することで、将来的に出現する可能性が高い変異株を予測するという方法です。このアプローチをオミクロンBA.2.86株に適用したところ、スパイクタンパク質の455、456、346番目の変異が適応度を上昇させると予測されました。実際に、その後出現し流行したBA.2.86の子孫株(JN.1、KP.2、KP.3株)は、これらの変異を獲得していたことが確認されました。この結果は、CoVFitが単一アミノ酸変異レベルでSARS-CoV-2の進化を予測可能であることを示唆しています(図3)。
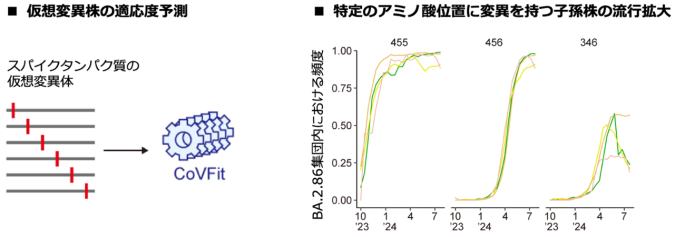
左)進化予測シミュレーションの方法。右)特定のアミノ酸位置に変異を持つBA.2.86子孫株の流行拡大。
本研究を通じて、SARS-CoV-2がどのように適応度を高めつつ進化し、流行拡大を繰り返してきたのかが明らかとなりました。また、CoVFitは今後、流行拡大リスクの高い変異株を超早期に同定するツールとして活用されることが期待されます。
現在、伊東准教授の研究チームは、ウイルスの進化と流行を理解・予測するためのAIおよびバイオインフォマティクス技術の開発に取り組んでいます。これらの技術は、ワクチン株のより効率的な選定法の確立等に応用でき、ウイルス感染症のより効率的な制御に貢献できると期待されます。
発表者・研究者等情報
東京大学医科学研究所 感染・免疫部門 システムウイルス学分野伊東 潤平 准教授
佐藤 佳 教授
Adam Strange
Wei Liu
Gustav Joas
Spyros Lytras
研究コンソーシアム「The Genotype to Phenotype Japan (G2P-Japan) Consortium」
論文情報
雑誌名 :Nature Communications題 名 :A Protein Language Model for Exploring Viral Fitness Landscapes
著者名 :Jumpei Ito*, Adam Strange, Wei Liu#, Gustav Joas#, Spyros Lytras, The Genotype to Phenotype Japan (G2P-Japan) Consortium, Kei Sato*
(#Equal contribution; *Corresponding author)
DOI: 10.1038/s41467-025-59422-w
URL: https://www.nature.com/articles/s41467-025-59422-w
研究助成
本研究は、伊東潤平准教授に対する科学技術振興機構(JST)「さきがけ(JPMJPR22R1)」、日本学術振興会(JSPS)「基盤研究B(25K00116)」および「若手研究(JP23K14526)」、ワクチン開発のための世界トップレベル研究開発拠点の形成事業(UTOPIA, 東京フラッグシップキャンパス(東京大学新世代感染症センター))「UTOPIA若手研究育成プログラム(P223fa627001、243fa627001h0003、253fa627001)」、シオノギ感染症研究進行財団「次世代育成支援研究助成金」、三菱UFJフィナンシャル・グループ「MUFJ-FGワクチン開発支援」、佐藤佳教授に対する日本医療研究開発機構(AMED)「医療分野国際科学技術共同研究開発推進事業 先端国際共同研究推進プログラム(ASPIRE)(パンデミックの 5W1H を理解するための研究)」、「新興・再興感染症に対する革新的医薬品等開発推進研究事業(重点感染症の病態発現と宿主の遺伝的背景の関連解析とその実証、ベトナムを拠点としたSARS-CoV-2 関連コウモリコロナウイルスの探索とそのウイルス学的特性の解明)」、AMED 先進的研究開発戦略センター(SCARDA)「ワクチン開発のための世界トップレベル研究開発拠点の形成事業(UTOPIA, 東京フラッグシップキャンパス(東京大学新世代感染症センター))、AMED SCARDA「ワクチン・新規モダリティ研究開発事業(100日でワクチンを提供可能にする革新的ワクチン評価システムの構築)」、日本学術振興会(JSPS)「国際共同研究加速基金(国際先導研究)(JP23K20041)」、JSPS 「基盤研究(A)(JP24H00607)」などの支援の下で実施されました。
用語解説
(注1)適応度(流行拡大能力)ある生物がどの程度多くの子孫を残せるかを示す進化生物学上の指標。ウイルスにおいては、感染拡大能力の目安として定義されることが多く、実効再生産数(1人の感染者が平均して何人に感染を広げるかを示す指標)により定量化されることが一般的である。適応度が高い変異株ほど、より多くの感染者を生み出す傾向がある。
(注2)タンパク質言語モデル
自然言語処理技術に基づいて構築された大規模言語モデルの一種で、単語や文の代わりにアミノ酸配列を学習対象とする。大量のタンパク質配列データを基に学習することで、タンパク質の構造的・機能的特徴を高次元の表現空間に埋め込む。これにより、タンパク質機能の予測、変異の影響評価、さらには新規タンパク質の設計など幅広い応用が可能となる。
(注3)ウイルスゲノム疫学データ
ウイルスのゲノム配列情報と、それに付随する疫学的メタデータ(採取日、採取場所、宿主情報など)を組み合わせたデータセット。SARS-CoV-2に関しては、これまでに2,000万件以上のウイルスゲノムが解読され、GISAIDデータベースに登録・公開されている。この情報は、ウイルスの進化、地理的拡散、流行株の同定・監視に不可欠である。
(注4)受容体結合ドメイン(RBD :Receptor Binding Domain)
スパイクタンパク質のS1サブユニットに存在し、SARS-CoV-2がヒト細胞へ侵入する際に必要な受容体ACE2と直接結合する機能を担う領域。RBDは中和抗体の主要な標的でもあり、この領域の変異は、ウイルスの感染性(ACE2との結合強度)や免疫逃避能力(中和抗体からの回避)に大きな影響を与える。したがって、RBDはウイルス進化やワクチン効果において極めて重要なドメインとされる。
問合せ先
〈研究に関する問合せ〉東京大学医科学研究所 感染・免疫部門 システムウイルス学分野
准教授 伊東 潤平(いとう じゅんぺい)
https://www.ims.u-tokyo.ac.jp/imsut/jp/lab/ggclink/section04.html
〈報道に関する問合せ〉
東京大学医科学研究所 プロジェクトコーディネーター室(広報)
https://www.ims.u-tokyo.ac.jp/