| 発表のポイント |
|---|
|
発表概要
新型コロナウイルスは、世界的流行のなかでゲノム変異を繰り返し、感染性が高まったり、ワクチンの効果を回避したりするような変異株の出現が懸念され新たな脅威になっています。どこでどのような変異株が感染拡大しているのか、新たな変異株が生じていないかを知ることは、変異株対策において極めて重要です。加えて、ウイルスゲノムの変異情報は、人から人への感染経路を把握する作業において大切な情報になります。感染経路がはっきりすれば、感染リスクが高い場所や行動を推測でき、感染を防ぐ可能性がある有効な対策に繋がります。2021年5月現在、世界中から集められた170万人以上の感染者から取得された新型コロナウイルスのゲノム配列が公開されています。こうした情報を活用するため、東大医科研附属ヒトゲノム解析センターは、IBMと共同でHGC SARS-CoV-2 Variant Browserを開発し、スーパーコンピュータSHIROKANE上で運用を開始しました。新型コロナウイルスのゲノム変異状況のモニタリングや懸念するべき変異株の迅速な発見、ゲノム変異情報を用いた感染経路の推定が可能になります。
発表内容
【研究の背景・課題】COVID-19の世界的な流行が続いており、日本国内も同様の状況が続いています。新型コロナウイルスは、流行の長期化に伴い新規のゲノム変異を蓄積し、時に新たな性質を獲得した変異株となり、各国の感染症対策に大きな影響を与えてきました。
一方で、こういったゲノム変異情報を活用することで、変異株よりも詳細なレベルで感染者の新型コロナウイルスが「どういった変異を持っており」「いつどの国から流入し」「どのように感染してきたのか」といった情報を得ることができます。このウイルスゲノム情報は、変異株の拡大状況や懸念するべき新たな変異株の出現を把握するための重要な情報となります。そのためには迅速に膨大な数の感染者から取得したウイルスゲノム情報を解析する必要があります。例えば、データベースGISAID(注1)には、2021年5月現在170万配列を超える新型コロナウイルスの配列が登録されています。このような膨大な数のウイルスゲノムを迅速に解析して結果を提供する体制が十分に整備できていませんでした。
特に、スポーツやコンサート、フェスティバルのようなマスギャザリングイベント(大規模集客イベント)については、COVID-19感染を拡大させる可能性から、対策の重要性がより増しています。欧州では、イギリス(注2)やオランダ(注3)などにおいてマスギャザリングイベントを行いながらそのリスクを評価する実証研究が進んでいます。また、日本においてもプロスポーツにおいて感染リスクを評価しながらの実施が試みられています。
今後、特に、プレイベントも始まっている東京オリンピック・パラリンピックが開催された場合には、国内の人流や海外から選手やスタッフ等の入国が増加することが想定されます。また、ワクチン接種による社会活動の回復も人流の増加要因となる一方で、ワクチン回避株の出現も懸念されています。従って、今後の変異株の状況把握は非常に重要です。更に、ウイルスゲノムの変異情報を用いると感染経路の把握にも繋がります。感染経路が判明すると感染リスクが高い場所や行動の同定に繋がり、有効な対策の立案が可能となります。
東大医科研附属ヒトゲノム解析センターとIBMは、新型コロナウイルスのゲノム変異状況のモニタリングならびに、ゲノム変異レベルでのウイルスの感染経路を把握できるHGC SARS-CoV-2 Variant Browserを開発することで、前述のボトルネックとなっていた課題に対応し、情報提供を行うことができる研究体制の確立を目指しました。
【今回の取り組み】
HGC SARS-CoV-2 Variant Browserは、IBMの基礎研究部門であるIBM ResearchがCOVID-19向けに開発してきたSARS-CoV-2 Variant Annotator(注4)および、IBMでデジタル変革を推進するIBM Garageと開発し公開したSARS-CoV-2 Variant Browser(注5)の技術をベースに開発を行い、東大医科研附属ヒトゲノム解析センターにて運用を開始しています。特に今回の取り組みでは、国外からの新型コロナウイルスの流入時期や国内の感染経路を把握するための新機能を、IBMと協力し実装しました。
また、東大医科研附属ヒトゲノム解析センターでは、新型コロナウイルスをシークエンス解析した生データをHGC SARS-CoV-2 Variant Browser で可視化できるよう解析するためのデータ解析パイプライン「HGC_CovidPipeLine」(注6)も合わせて公開しました。HGC SARS-CoV-2 Variant Browserを活用することで、下記のようなウイルスゲノム分析・変異株モニタリングを可能にしています。
<海外からの流入時期ならびに感染経路推定>
感染者から採取した新型コロナウイルスが、過去に報告された新型コロナウイルスと比べて、どの時期に海外から国内に流入したかどうかを調べることができます。また国内に流入した後に、どの地域にて報告があったかも確認をすることができるため、一部の地域に留まっているのか、他の地域でも広がっているのかを検討することができます (図1)。
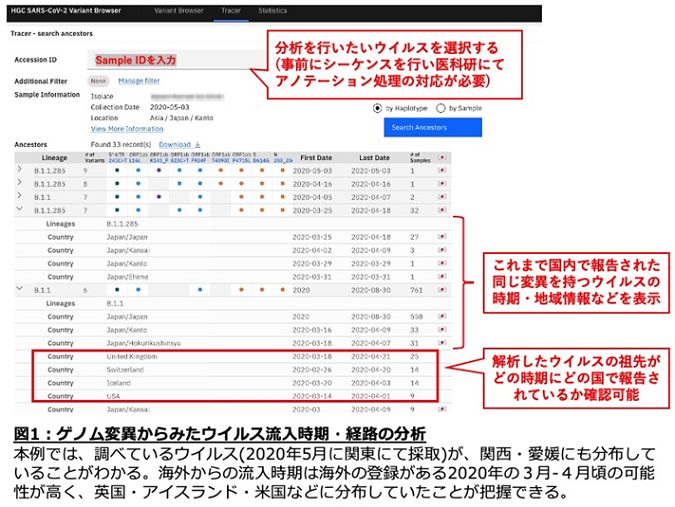
<新型コロナウイルスの変異状況の把握>
国内で新たな変異が発見された場合に、海外でこれまでに同様の報告があるのかどうかを調べることができます。変異での検索だけでなく、地域や時期などで検索をかけることができ、その検索条件にあった、変異の状況を簡便に俯瞰できます (図2)。
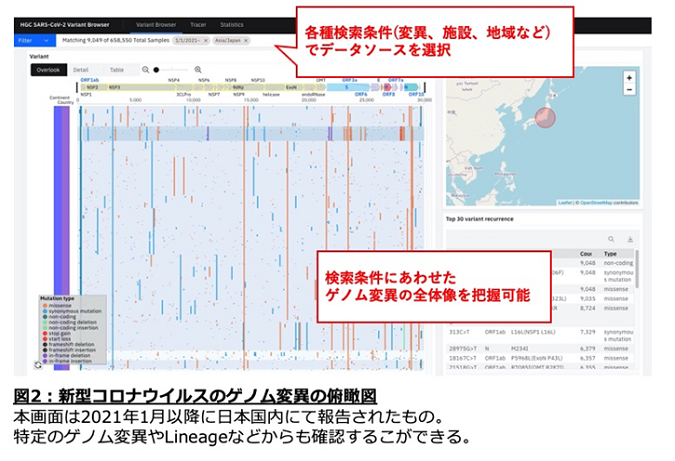
<新型コロナウイルスのゲノムの国内登録状況・動向>
今回のシステムの解析にあたり、最も大切なのは過去に登録された新型コロナウイルスのゲノムデータの活用です。公開されているゲノムデータについは、国によっても差があり、また国内においても都道府県ごとに違いがあります。こうしたそれぞれのデータの傾向を把握するために、本システムで活用している世界・日本の公開されている新型コロナウイルスゲノムの登録数や変異株などを一目でモニタリングできるビューアーも実装しています (図3)。
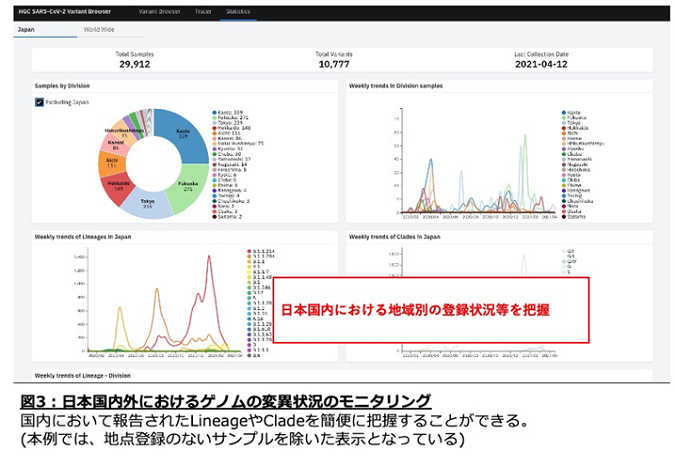
【今後の取り組み】
新型コロナウイルスは、今後も新たなゲノム変異を蓄積していくため、国内外でワクチン回避を起こす恐れのある変異株や、新たな動物感染を引き起こす変異株の出現が懸念されます。本取り組みを通じて、こうした課題に引き続き対応を進めていきます。また、より早く、懸念される変異を捉えるために様々な団体や地方自治体などとも協力しゲノムデータ活用できる体制を整えていきます。
東大医科研附属ヒトゲノム解析センターは、これまでに蓄積したゲノム研究やデータサイエンス研究の知見に基づき、新型コロナウイルス感染症患者のゲノム情報を解析し新たな治療法に繋がる研究を進めてきました。今回のシステムをコロナ制圧タスクフォースにおいて収集する新型コロナウイルスゲノムの解析やオリンピック・パラリンピックによる人流に起因するウイル伝播のモニタリングに活用し、新型コロナウイルスの基礎研究ならびに感染拡大防止策の立案に貢献して行けると考えています。
IBMは、これまでCOVID-19への様々な取り組みを行ってきました。今回、東大医科研附属ヒトゲノム解析センターに提供したVariant Annotator および可視化をするVariant Browserは、IBMがCOVID-19の流行初期からゲノム変異に着目し研究開発の取り組みを行ってきたものです。また、日本向けにも国内にて公開された新型コロナウイルスゲノムを解析し、解析結果を公開してきています(注7)。IBMにおいても、IT技術を活用した取り組みを通じて、引き続きCOVID-19への取り組みを継続する予定です。
問い合わせ先
<内容に関するお問い合わせ先 >東京大学医科学研究所附属ヒトゲノム解析センター
センター長・教授 井元 清哉(いもと せいや)
http://www.ims.u-tokyo.ac.jp/imsut/jp/
<報道に関するお問い合わせ先 >
東京大学医科学研究所 国際学術連携室(広報)[担当:清水]
https://www.ims.u-tokyo.ac.jp/imsut/jp/
日本アイ・ビー・エム株式会社 (広報) [担当:安原・下岡]
Email: PRESSREL@jp.ibm.com
用語解説・参考文献:
(注1)GISAID: https://www.gisaid.org/
(注2)The United Kingdom Government. Information on the Events Research Programme. 2021: https://www.gov.uk/government/publications/guidance-about-the-events-research-programme-erp-paving-the-way-for-larger-audiences-to-attend-sport-theatre-and-gigs-safely-this-summer/guidance-on-the-events-research-programme
(注3)de Vrieze J. Dutch studies bring back the fun—but are they good science? Science 2021; 372(6541): 447
(注4)Variant Annotator: Koyama T., Platt D., Parida L. “Variant analysis of SARS-CoV-2 genomes.” Bull World Health Organ. 2020;98:495-504.
(注5)SARS-CoV-2 Variant Browser - IBM Functional Genomics Platform: https://ibm.biz/functional-genomics
(注6)HGC_CovidPipeLine: https://www.hgc.jp/~ppl_ht/CovidPipeLine/latest/docs/
(注7)Tokumasu R., Weeraratne D., Snowdon J., Parida L., Kudo M., Koyama T. “Introductions and evolutions of SARS-CoV-2 strains in Japan” medRxiv 2021.02.26.21252555.
