| 発表のポイント |
|---|
|
概要
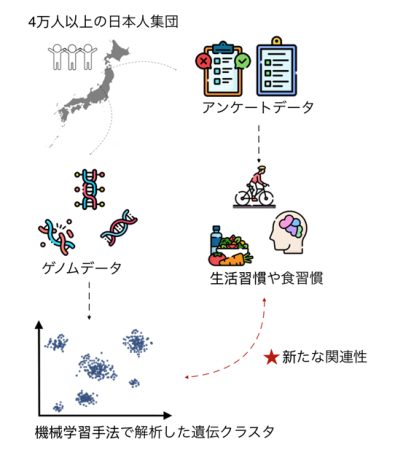
東京大学医科学研究所健康医療インテリジェンス分野の井元清哉教授、Chen Yichi特任研究員、同研究所シークエンスデータ情報処理分野の片山琴絵准教授、株式会社ディー・エヌ・エーの子会社である株式会社アルムの石田幸子博士らの研究グループは、共同研究によって4万人以上の大規模遺伝子データを解析し、日本人集団内の微細な遺伝構造と食習慣・生活習慣との関連を明らかにしました。本研究では、ゲノム研究プロジェクト「MYCODE Research」(注1)の下で収集された遺伝子データを対象に、PCA(注2)、UMAP(注3)、DBSCAN(注4)といった機械学習手法を用いて微細な遺伝構造の抽出を試みました。その結果、比較的均一と言われている日本人集団内にも複数の確かな遺伝クラスタが存在し、これらは祖先の地理的ルーツと関連していることが示されました。また、分類された遺伝クラスタは、HDLコレステロールや肝機能、血糖コントロールなどの特定の形質に関わる遺伝子群に有意な違いがあること、さらには、野菜や牛乳の摂取頻度、睡眠の質などの食習慣や健康行動に違いがあることが示されました(図1)。
本研究成果は、2025年7月12日(英国時間)に、国際科学雑誌『Communications Biology』にオンライン掲載されました。
発表内容
〈研究の背景〉近年、個人の遺伝子データを詳しく分析し、病気の予防や健康管理に役立てる取り組みが広がっています。特に、機械学習や人工知能による新しい解析技術の発展により、疾患などの個人の属性をあらかじめ与えることなく膨大なデータの中から類似性を見つけ出す「教師なし学習」(注5)が、新たな特徴を共有する集団の発見において注目されています。
これまで日本人は遺伝的に均質な集団と考えられてきましたが、最新の機械学習解析により、同じ集団内にもわずかな遺伝的違いが存在することが分かってきました。こうした違いは、地域ごとの祖先構成や歴史的移動と関わっている可能性があり、体質や病気のかかりやすさにも影響を与えると考えられています。しかし、こうした微細な遺伝構造と、日々の食習慣や生活習慣との関連性は十分に解明されていませんでした。
〈研究内容と成果〉
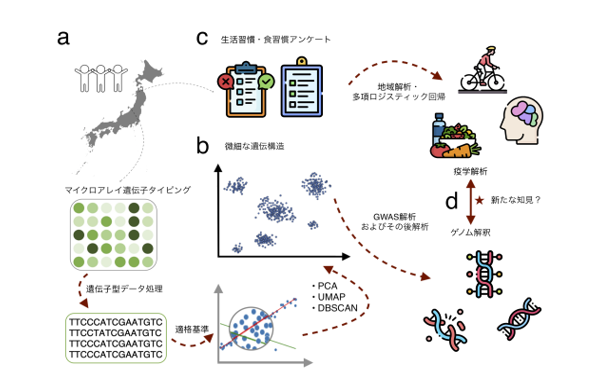
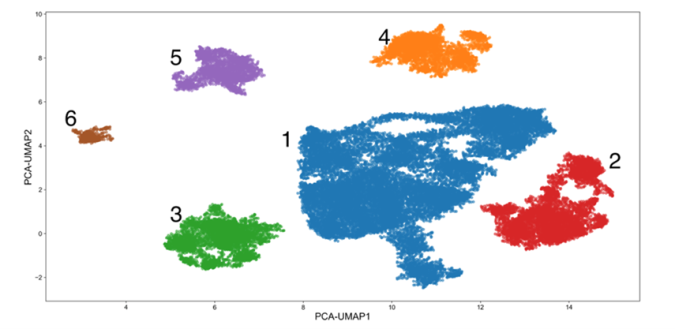
本研究では、株式会社DeNAライフサイエンスが提供した個人向け遺伝子検査サービス「MYCODE」(注1※)を利用した会員のうち、研究への参加について同意された会員4万人以上の日本人の大規模遺伝子データを対象に、PCA、UMAP、DBSCANといった機械学習手法を組み合わせ、日本人集団内の微細な遺伝構造を解析しました(図2)。解析の結果、遺伝的違いによって解析対象の集団は6つの遺伝クラスタに分かれました。中でもクラスタ2(赤、15.2%)とクラスタ3(緑、11.2%)は最も異なる特徴を示し、クラスタ1(青、54.5%)は集団全体を代表する特徴を持ちながら、クラスタ5(紫、7.8%)やクラスタ6(茶、1.8%)と特に近い遺伝的特徴を共有していました。なお、クラスタ5と6は人数が少なかったため、解析ではクラスタ1とまとめて「クラスタ1*」として扱い、全体を4つのクラスタに分けて解析を行いました。
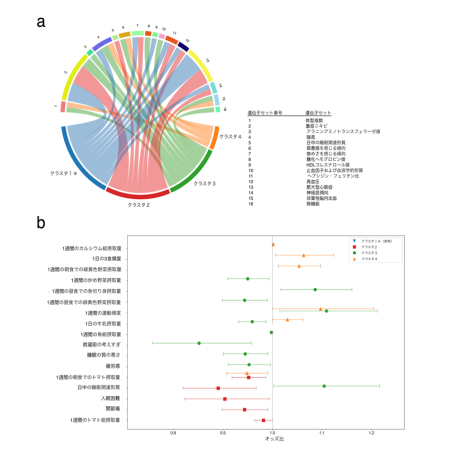
さらに、年齢・性別・BMIを考慮した統計解析により、食習慣や生活習慣との関連を調べたところ、175項目のうち21項目で有意な関連が認められました(図3b)。例えば、クラスタ3とクラスタ4では野菜を食べる頻度が高く、クラスタ3では牛乳を飲む頻度も高いなど、各クラスタに特徴的な食習慣や生活習慣が明らかになりました。
発表者・研究者等情報
東京大学医科学研究所 附属ヒトゲノム解析センター 健康医療インテリジェンス分野
井元 清哉 教授
Chen Yichi 特任研究員
シークエンスデータ情報処理分野
片山琴絵 准教授
株式会社アルム
石田幸子
論文情報
雑誌名:Communications Biology題 名:Intricate interactions between fine-scale genetic structure, lifestyle, and dietary habits in the Japanese population
著者名:Yichi Chen, Kotoe Katayama, Sachiko Ishida, Seiya Imoto※(※責任著者)
DOI: 10.1038/s42003-025-08479-w
URL: https://www.nature.com/articles/s42003-025-08479-w
研究助成
本研究は、株式会社DeNAライフサイエンスとの共同研究として実施されました。
用語解説
(注1)MYCODE Research株式会社DeNAライフサイエンスが提供していた個人向け遺伝子検査サービス「MYCODE」を利用した会員のうち、データの研究利用に同意いただいた方を対象としたユーザー参加型のゲノム研究プロジェクト。約9割の会員が研究同意され、これまでに累計約40件のアカデミアや企業との共同研究が行われてきた 。
※ 現在「MYCODE」はサービスを終了し、株式会社DeNAライフサイエンスの研究事業である「MYCODE Research」は、株式会社ディー・エヌ・エーの子会社である株式会社アルムに事業承継されている。
(注2)PCA(主成分分析)
遺伝子データのような高次元データを統計的に整理し、データのバラツキを最もよく説明する重要な特徴(主成分)を抽出することで、どのような変数(遺伝子)がデータのバラツキに寄与しているかを解析することができるデータ解析手法。
(注3)UMAP
「Uniform Manifold Approximation and Projection」の略。複雑な高次元データの構造を二次元や三次元にわかりやすく可視化できる解析手法。
(注4)DBSCAN
「Density-Based Spatial Clustering of Applications with Noise」の略。データを類似性によってまとめ、クラスタに分類する手法。
(注5)教師なし学習
機械学習手法のひとつ。例えば、疾患である、疾患でないというような個人の属性を学習データとして与えることなく、個人を特徴付ける例えば検診などのデータのみから個人のグルーピングを行う方法。
(注6)遺伝子セット解析(Gene Set Enrichment Analysis, GSEA)
事前に定義した注目している遺伝子セットに対して、特定の機能を有する遺伝子や特定の疾患に関連する遺伝子が有意に含まれているかどうかを解析するデータ解析手法。
問合せ先
〈研究に関する問合せ〉東京大学医科学研究所 附属ヒトゲノム解析センター 健康医療インテリジェンス分野
教授 井元 清哉(いもと せいや)
https://www.ims.u-tokyo.ac.jp/imsut/jp/lab/hgclink/page_00072.html
〈報道に関する問合せ〉
東京大学医科学研究所 プロジェクトコーディネーター室(広報)
https://www.ims.u-tokyo.ac.jp/