概要
新型コロナウイルス(SARS-CoV-2)「オミクロン株」は次々と変異を繰り返して流行の波を形成しています。京都大学医生物学研究所の橋口隆生教授と同大薬学研究科 矢島久乃博士課程学生、北海道大学大学院薬学研究院の前仲勝実教授と同大学大学院生命科学院 安楽佑樹博士課程学生、東京大学医科学研究所システムウイルス学分野の佐藤佳教授らの研究グループは、昨年流行したXBB系統から30箇所以上の変異をSpike蛋白質に獲得し、新たな変異株として今年流行の大きな波を形成したBA.2.86とJN.1のSpike蛋白質の構造をクライオ電子顕微鏡を用いて解明しました。現在流行中の変異株であるKP.3などはこの子孫系統になります。構造的な特徴として、ウイルスが細胞に感染する際に最初に細胞の入口として利用する受容体ACE2に対して、これまでに観測されたことのない様式でもウイルスが受容体に結合できる様子(中間体構造)が観察されました。さらに、獲得した変異のうち、K356Tという変異が中和抗体からの逃避能を高めていることが分かりました。
新しい変異株が感染力を高めたり、抗体から逃避する変異を獲得した理由を説明できる構造情報が得られたことで、病原性の理解と変異株対応ワクチン開発などの創薬研究への進展が期待されます。一連の構造情報は、国際的な構造データベースであるProtein Data Bank(PDB)とElectron Microscopy Data Bank(EMDB) を通じて世界中の研究者が無償で利活用することができます。
本研究成果は、2024年10月7日に、国際学術誌「Nature Communications」にオンライン掲載されました。
1.背景
新型コロナウイルス(SARS-CoV-2)「オミクロン株」は次々と変異を繰り返して流行の波を形成しています。特に、ウイルス粒子の表面に存在するSpike蛋白質(注1)は、世界的な流行を引き起こす変異株ごとに毎回変異を獲得し、ウイルスが細胞に感染する際に必要なACE2受容体(注2)との結合する能力やヒトの免疫系から逃れる能力が変化することで、新たな感染流行の波を形成する際に最も重要な役割を果たしています。すなわち、Spike蛋白質は、SARS-CoV-2が病原性を発揮するために必須の分子であると同時に、我々ヒトの獲得免疫系がウイルスを認識して、ウイルス排除のための液性免疫(注3)や細胞性免疫(注4)などの免疫応答を起こすためのワクチン抗原(注5)そのものでもあります。従って、新たに出現した変異株のSpike蛋白質の構造や性状を理解することは、病原性の理解とともにワクチン開発や創薬研究にも役立ちます。
本研究では、京都大学医生物学研究所の橋口隆生教授、同大薬学研究科 矢島久乃博士課程学生、北海道大学大学院薬学研究院の前仲勝実教授、同大学大学院生命科学院の安楽佑樹博士課程学生、東京大学医科学研究所システムウイルス学分野の佐藤佳教授らの研究グループが、昨年大きな流行の波を形成したXBB系統から30箇所以上の変異をSpike蛋白質に獲得し、新たな変異株として今年、流行の大きな波を形成したBA.2.86及びJN.1系統のSpike蛋白質の構造特性を解明しました。現在流行中の変異株であるKP.3などはこの子孫系統になります。
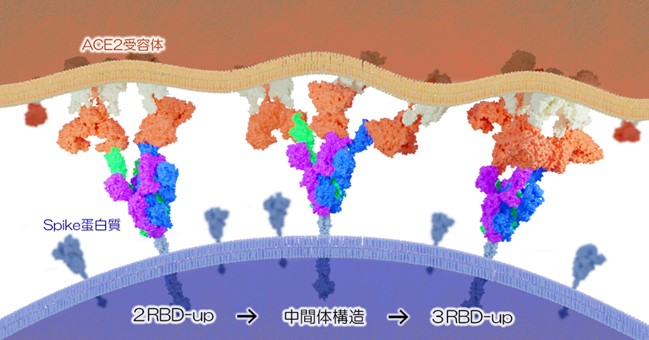
2.研究手法・成果
本研究チームは、ウイルスを構成する蛋白質の形(構造)を原子レベルで可視化することができるクライオ電子顕微鏡(注6)という最先端の顕微鏡を用いて本研究成果を挙げました。クライオ電子顕微鏡による解析の結果、新たな変異株であるBA.2.86及びJN.1系統のSpike蛋白質を用いて、これまで観測されなかった中間体構造の観測に成功しました。以前の研究では、SARS-CoV-2のSpike蛋白質のうちACE2受容体結合に重要な受容体結合ドメイン(RBD)という部分が“UP”構造のときのみACE2受容体に結合した構造が報告されていました。しかし、BA.2.86及びJN.1は、RBDの可動性を高めることで、RBDが“DOWN”状態でもACE2受容体に結合する構造特性を示しました(動画)。こうした変化はBA.2.86及びJN.1による感染力に寄与していると考えられます。また、シュードウイルス(注7)を用いた中和試験(注8)により、ウイルスが獲得した変異のうち、K356Tという変異が中和抗体からの逃避能を高めていることも分かりました。以上の成果から、BA.2.86及びJN.1系統のSpike蛋白質に生じた変異の意義や中間体構造が明らかとなり、病原性の理解や創薬研究の基盤となる構造情報が得られました。この構造情報は、国際的な構造データベースであるProtein Data Bank(PDB)とElectron Microscopy Data Bank(EMDB) を通じて世界中の研究者が無償で利活用することができます。
3.波及効果、今後の予定
本研究チームは、数カ月ごとに出現するSARS-CoV-2変異株に迅速対応するために、「The Genotype to Phenotype Japan(G2P-Japan) :ウイルスの性状解析を行うコンソーシアム」及び「Japanese Consortium on Structural Virology(JX-Vir) :ウイルスの構造解析を行うコンソーシアム」という2つのコンソーシアムを連携させた研究をこれまでも継続的に実施してきました。一つ一つの研究室でできることには限界がありますが、研究者間のネットワークを密にしてコンソーシアム型研究を行うことで、感染症有事にも平時にも迅速かつ高精度・高情報量の研究を実施する体制を構築しています。今後も社会的に影響の大きい感染症研究に取り組んでいく予定です。
4.研究プロジェクトについて
本研究は、橋口隆生教授らに対する日本医療研究開発機構(AMED)「先端国際共同研究推進プログラム(ASPIRE)(パンデミックの 5W1H を理解するための研究)」、AMED 先進的研究開発戦略センター(SCARDA)「ワクチン開発のための世界トップレベル研究開発拠点の形成事業(ヒト免疫に関する京都大学サポート機関)日本学術振興会 研究拠点形成事業(A.先端拠点形成型)(ウイルスの二面性の理解・活用のための国際研究拠点形成)などの支援の下で実施されました。
用語解説
論文タイトルと著者
タイトル:Structural basis for receptor-binding domain mobility of the spike in SARS-CoV-2 BA.2.86 and JN.1.(SARS-CoV-2 BA.2.86及びJN.1のSpike蛋白質の受容体結合ドメインの可動性に関する構造基盤)著 者:矢島久乃、安楽佑樹、郭悠、木村(寺角) 香菜子、Arnon PLIANCHAISUK、奥村佳穂、名倉淑子、新勇介、逸見拓矢、黒田大祐、高橋宜聖、喜多俊介、佐々木慈英、澄田裕美、The Genotype to Phenotype Japan (G2P-Japan)、伊東 潤平、前仲勝実、佐藤佳、橋口隆生
掲 載 誌:Nature Communications
DOI:10.1038/s41467-024-52808-2
URL : https://www.nature.com/articles/s41467-024-52808-2
問い合わせ先
<研究に関するお問い合わせ先> 橋口隆生(はしぐち たかお)
京都大学医生物学研究所・教授
https://kdb.iimc.kyoto-u.ac.jp/profile/ja.f236b4995e0e922e.html
<報道に関するお問い合わせ先>
京都大学 渉外・産官学連携部広報課国際広報室
https://www.kyoto-u.ac.jp/ja
北海道大学 社会共創部広報課
https://www.hokudai.ac.jp/
東京大学医科学研究所 プロジェクトコーディネーター室(広報)
https://www.ims.u-tokyo.ac.jp/
