| 新型コロナウイルス(SARS-CoV-2:注1)感染症(COVID-19:注2)の脅威に立ち向かうための共同研究グループ「コロナ制圧タスクフォース」では、これまでCOVID-19重症化因子の解明等重要な知見を提示してきました(参考1、参考2)。今回、王青波准教授(東京大学大学院医学系研究科遺伝情報学/研究当時)、岡田随象教授(大阪大学大学院医学系研究科遺伝統計学/東京大学大学院医学系研究科遺伝情報学/理化学研究所生命医科学研究センターシステム遺伝学チーム)、南宮湖専任講師(慶應義塾大学医学部感染症学教室)を中心とした研究グループは、COVID-19の患者1,405名の検体を用いた血漿中タンパク質の網羅的な解析により、血漿タンパク質量の個人差に寄与するヒトゲノム配列(注3)を大規模に同定しました。 本研究では、これまでの先行研究において原因変異として精緻に推定(fine-mapping:注4)され、血漿タンパク質発現を制御する582箇所のヒトゲノム変異を比較し、タンパク質の機能情報を統合的に解析することにより、原因変異においてはミスセンス変異や機能喪失変異(注5)である確率が1000倍以上高いことが示されました。また、血液中mRNA発現量との比較や、疾患等の複雑形質との関連の評価、COVID-19重症度との相互作用の解析により、血漿におけるタンパク質発現を決定する多様な要素を明らかにすると共に、mRNA発現のみでは同定できない疾患との関連性が広く存在することが示されました。これらの結果は、ゲノムの個人差による遺伝子発現制御を通じた複雑形質の関連について、より詳細な理解につながると考えられます。 本研究成果は、2024年9月24日(英国時間)に国際科学誌Nature Geneticsオンライン版に掲載されました。コロナ制圧タスクフォースは未来のパンデミックに備える社会の公器として、引き続き活動を続けていきます。 |
1.研究の背景と概要
個体間でのヒトゲノム配列の違いにより、身長や疾患等の形質(生物が持つ性質や特徴のこと)だけでなく、様々な遺伝子発現量そのものも個体差が生じることが知られています(参考2)。遺伝子は体内で発現するタンパク質を規定するDNA配列の基本単位である一方、体内において多くのタンパク質量を直接大規模に測定することは技術的に困難でした。そのため、ヒトゲノムを解析する集団遺伝学の分野における多くの研究では、遺伝子発現量の指標としてメッセンジャーRNA (mRNA)発現量が用いられてきました。しかし、その指標であるmRNA量は転写後修飾や翻訳等のプロセスを経た最終産物であるタンパク質の量を正確に反映するわけではないことが知られており(注6)、タンパク質量の粗い「近似」としての弱点が指摘されていました。i)血漿タンパク質発現を制御するゲノム上原因変異の網羅的同定
今回、本研究グループは、COVID-19 患者1,405名のうち1,369名の血液細胞を用いて約3,000のタンパク質の血漿におけるmRNA発現量と、ヒトゲノム配列の個人差との関連を解析しました。今回利用されたOlink Proximity Extension Assayと呼ばれる最新の技術に基づくタンパク質発現量データの網羅的な解析は、これまで東アジア人集団の研究では限られていました。
コロナ制圧タスクフォースの解析結果を、欧米での先行研究(UK Biobank = UKB, Plasma Proteome Project)と比較することで、血漿タンパク質を制御する確率の推定値(Posterior Inclusion Probability = PIP)が十分に高い変異(putative causal variant = 原因変異)においては制御効果の大きさ(効果量)が先行研究と高精度で一致すること(図1 A、横軸)、また集団の遺伝的背景(注7)が近い場合に効果量の一致度が特に大きいことを見出しました(図1 A、縦軸)。また各変異の機能情報を解析することで、特にミスセンス変異や機能喪失変異(注5)等のタンパク質コーディング領域に原因変異が生じる確率が高いこと(図1B、 右)、さらにタンパク質を直接コードしないmRNAの発現調節機能に関する情報も原因変異である確率を高める一因であること(図1B、中央)を明らかにしました。
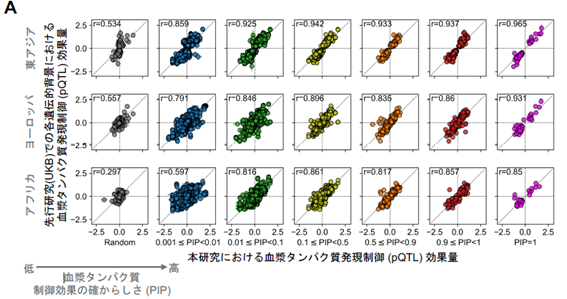
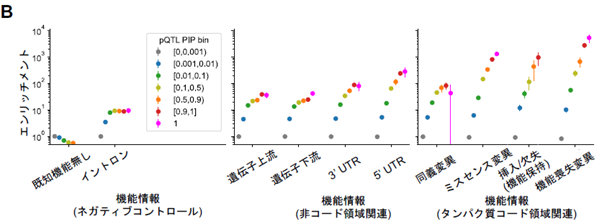
【図 1】今回同定された血漿タンパク質発現制御に関わるゲノム多型(pQTL)は、先行研究の結果とよく一致し(A)、種々の機能情報のエンリッチメントが見られた(B)。
ii)血漿タンパク質mRNA発現と血液中mRNA発現の一致/不一致要因の解明
今回の研究において対象となった1,405名のうち1,019名は従来のRNA-seqを用いた血液中mRNA発現量も測定されており、998名の患者においては、血液中mRNA発現量と血漿タンパク質mRNA発現量の両方のデータを有していました。そこで、本研究グループでは両者のデータの比較解析を実施しました。
まず、先行研究で報告されている体内全体でのmRNA発現量のうち、血液中mRNA発現の割合に着目したところ、体内全体のうち血液中mRNA発現の割合が低い遺伝子では、特に血液中mRNA-タンパク質mRNA間での相関が低いことを見出しました(図2A)。この解析結果により、血漿タンパク質mRNA発現を構成する要素として血液細胞由来のmRNAの翻訳以外に多様な組織由来のタンパク質が大きな割合を占めることが示唆されました。一方、ゲノム変異単位ごとに解析すると、該当する変異が血液中mRNA発現と血漿タンパク質mRNA発現の双方に関して原因変異となる場合には、両者の効果量は高い精度で一致することを見出しました(図2B、右下)。本解析結果はmRNAとタンパク質の制御に関して一定数の共通機構が存在することを示す結果と考えられました。
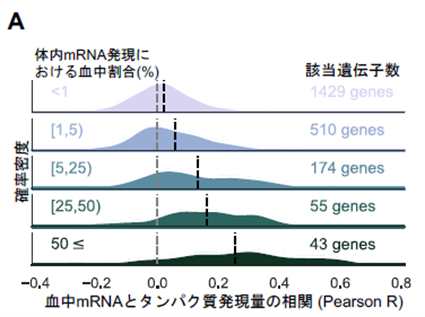
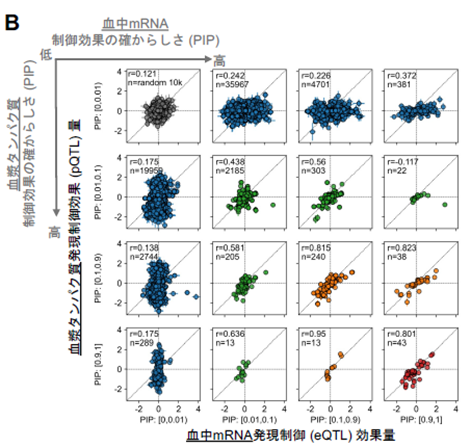
【図 2】遺伝子の mRNA とタンパク質発現量の相関において、体内全体における血液中mRNA 発現割合が寄与することが示された(A)。また、統計的 fine-mapping を行うことにより、ゲノム多型が血液中 mRNA 発現を制御する効果(eQTL 効果)と、血漿タンパク質発現を制御する効果(pQTL 効果)は、概ねよく相関することが示された(B)。
iii)血中での遺伝子発現制御機構に基づく、複雑形質や疾患の再理解
ゲノム多型が血液中mRNAを制御する効果(eQTL効果)と血漿タンパク質mRNAを制御する効果(pQTL効果)をそれぞれ分類して解析することにより、既存のゲノム多型と疾患を含む多くの複雑形質の相関をセントラルドグマ(注6)の各段階に焦点を当てて再解釈することが可能となりました。例として、BioBank Japanデータが有する種々の形質情報に関して、ゲノム変異が遺伝子をどの段階で制御するかを網羅的に解析した結果(図3)から、APOE遺伝子はmRNAレベルではなくタンパク質レベルの制御を受けて糖尿病リスクに寄与することが示唆されました。こうしたセントラルドグマの各段階を加味した精緻な理解は、生物学的機序における特定の段階をターゲットとする創薬アプローチの可能性を広げる可能性があります。
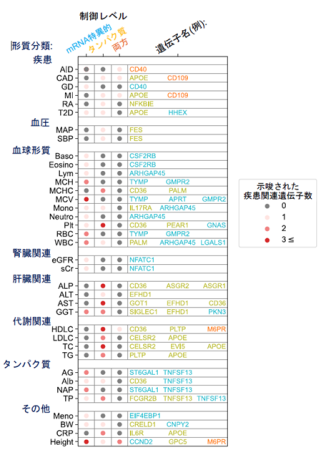
【図3】BioBank Japanデータにおける種々の形質に関して、ゲノム多型が血液中において関連遺伝子を主にmRNAまたはタンパク質どちらの段階で制御をするかが網羅的に推定された。
iv)相互作用を加味したさらに精緻な制御機構の理解
遺伝子発現量の増減が疾患リスクに寄与するだけではなく、疾患進行度に応じて遺伝子発現量も変化することが知られています。今回の研究ではCOVID-19重症度と遺伝子発現量の増減を、従来のmRNA発現に加えタンパク質をコードするmRNA発現も併せて評価することにより、疾患と遺伝子発現の相互作用において、mRNAとタンパク質で異なるプロファイリングが必要であることが示唆されました。一例として、図4に示すIL1RL1遺伝子では、重症及び最重症患者ではmRNAとタンパク質の両者の発現量がよく一致しており、COVID-19重症化による免疫応答が動的に転写を促進すると考えられました。
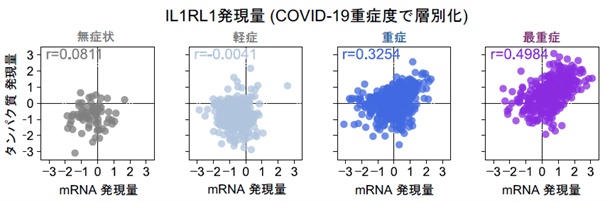
【図4】COVID-19重症度により、血液中mRNA発現量と血漿タンパク質mRNA発現量の相関が大きく変化する遺伝子の一例(IL1RL1)。
2.研究の成果と意義・今後の展開
今回の研究成果により、種々の血漿タンパク質mRNA量の個人差に寄与するヒトゲノム配列が大規模に同定されました。また、従来のmRNA発現量と比較することにより、個々のヒトゲノム配列に関して、遺伝子発現量の制御がセントラルドグマのどの段階で起こるのかがより精緻に明らかになりました。多種多様な転写/翻訳制御が疾患リスクと関連して相互作用をすることが示され、体内のmRNAのみならずタンパク質をコードするmRNA量を大規模に測定することの重要性が示唆されました。今回の研究結果は、遺伝子発現制御の仕組みのより詳細な理解の第一歩となると考えられます。同時に、血漿タンパク質mRNA量は体内の多様な組織で生じる生命活動のごく一部を反映したものであること、今回の解析において全てのタンパク質が網羅されているわけではないこと、タンパク質の測定技術自体が発展途上であり測定限界やバイアスが存在しうることが留意されます。より一般的な変異効果を予測するための機械学習的手法の開発や、複数手法・組織での複合的解析が今後の展開として期待されます。
3.論文
英文タイトル:Statistically and functionally fine-mapped blood e/pQTLs from 1,405
humans reveal their distinct regulation patterns and disease relevance
タイトル和訳:1,405人の統計と機能解析により精緻に推定された血液e/pQTLが、異なる制御パターンと疾患への関連を明らかにする
著者名:Wang, Q.S. et al.
掲載誌:Nature Genetics
DOI:10.1038/s41588-024-01896-3
URL:https://www.nature.com/articles/s41588-024-01896-3
humans reveal their distinct regulation patterns and disease relevance
タイトル和訳:1,405人の統計と機能解析により精緻に推定された血液e/pQTLが、異なる制御パターンと疾患への関連を明らかにする
著者名:Wang, Q.S. et al.
掲載誌:Nature Genetics
DOI:10.1038/s41588-024-01896-3
URL:https://www.nature.com/articles/s41588-024-01896-3
4.特記事項
本研究は、日本医療研究開発機構(AMED)創薬支援推進事業「新型コロナウイルス感染症(COVID-19)に対するワクチン開発」「新型コロナウイルス感染症の遺伝学的知見に基づいた分子ニードルCOVID-19粘膜免疫ワクチンの開発」、新興・再興感染症に対する革新的医薬品等開発推進研究事業「新型コロナウイルス感染症の重症化阻止を目指した医薬品・次世代型ワクチン開発に必要な遺伝学・免疫学・代謝学的基盤研究の推進」、新興・再興感染症に対する革新的医薬品等開発推進研究事業「新型コロナ変異ウイルスに対する遺伝学的、免疫学的、代謝学的病態解明および治療戦略の策定」、新興・再興感染症研究基盤創生事業(多分野融合研究領域)「新型コロナウイルス感染症後遺症の病態生理の多分野融合による解明」、ゲノム創薬基盤推進研究事業「大規模集団ゲノムデータを利用した遺伝子発現制御文法の機械学習による、VUS病原性の網羅的評価と実験検証」、AMED SCARDA「ワクチン開発のための世界トップレベル研究開発拠点の形成事業」、科学技術振興機構(JST) 戦略的創造研究推進事 CREST「先端ゲノム解析と人工知能によるコロナ制圧研究」(JPMJCR20H2)、戦略的創造研究推進事業 個人型研究さきがけ「パンデミックに対してレジリエントな研究体制構築のための基盤研究」(JPMJPR21R7)、創発的研究支援事業「ゲノム制御機構を解明する、解釈可能な汎用予測モデルの構築」(JPMJFR225Y)、大阪大学免疫学フロンティア研究センター(IFReC)、大阪大学感染症総合教育研究拠点(CiDER)の支援を受けました。また、イーパーセル株式会社の北野譲治様を始めとする本研究プロジェクトにご賛同いただいた寄付者からの寄付金を基に実施されました。データ解析において東京大学医科学研究所ヒトゲノム解析センターのスーパーコンピュータ「SHIROKANE」を利用しました。
5.本プロジェクトに参画したタスクフォースメンバー
慶應義塾大学医学部:教授 福永興壱、教授 金井隆典、専任講師 南宮湖東京医科歯科大学:M&Dデータ科学センター長 宮野悟、教授 小池竜司、名誉教授 木村彰方
東京大学大学院医学系研究科:准教授 王青波(研究当時)、教授 岡田随象(大阪大学大学院医学系研究科、理化学研究所生命医科学研究センター)
東京大学医科学研究所附属ヒトゲノム解析センター:教授 井元清哉
京都大学大学院医学研究科:教授 小川誠司
北里大学:教授 高野友美
名古屋大学大学院医学系研究科:教授 石井誠(研究当時 慶應義塾大学医学部:准教授)
6.コロナ制圧タスクフォースに参加する全国の医療機関
詳細は、コロナ制圧タスクフォースHP(参考2)をご参照ください。【参考】
(参考1)2022年8月9日プレスリリース
「コロナ制圧タスクフォース」
COVID-19疾患感受性遺伝子DOCK2の重症化機序を解明
-アジア最大のバイオレポジトリーでCOVID-19の治療標的を発見-
https://www.keio.ac.jp/ja/press-releases/2022/8/9/28-126102/
(参考2)2022年8月23日プレスリリース
「コロナ制圧タスクフォース」
COVID-19 患者由来の血液細胞における遺伝子発現の網羅的解析
-重症度に応じた遺伝子発現の変化には、ヒトゲノム配列の個人差が影響する-
https://www.keio.ac.jp/ja/press-releases/2022/8/23/28-126399/
用語解説
(注1)新型コロナウイルス(SARS-CoV-2):新型コロナウイルス感染症の原因となるウイルスのことを表します。2002年に流行したSARSコロナウイルスとウイルスが似ているため、SARS-CoV-2と命名されました。(注2)新型コロナウイルス感染症(COVID-19):coronavirus disease 2019(2019年に発生した新型コロナウイルス感染症)を略した言葉で、新型コロナウイルスによる病気のことを表します。2019年の終わり頃に、中国・武漢を中心に発生したのを皮切りに、その後、世界中に感染が拡大しました。新型コロナウイルスに感染すると、発熱や咳、息苦しさといった症状が出て、感染が肺に及び肺炎を発症すると呼吸困難に陥ります。
(注3)ヒトゲノム配列:遺伝子とは、親から子へ体を作るときの特徴を伝えるための1つ1つの情報のことで、生命現象の中で重要な働きをするタンパク質の設計図です。ゲノムとは、使わない情報も含んだすべての遺伝情報のことです。ヒトゲノム配列には個人差が存在することが知られ、ゲノム変異と呼ばれます。
(注4)fine-mapping:一般に形質に関連するゲノム変異は複数存在し、そのうち原因となる変異は少数であることがわかっています。多数の関連する変異から原因である可能性が最も高い変異群を統計的に推定することを(statistical)fine-mappingと呼びます。
(注5)ミスセンス変異/機能喪失変異:ゲノム変異のうち、タンパク質を構成するアミノ酸一つに変化が生じるものはミスセンス変異、タンパク質の翻訳の停止や、フレームシフト(DNAの塩基配列3つで1つのアミノ酸をコードするが、その3つで1組の読み取りの枠がずれること)による大規模なアミノ酸配列の変化等を生じるものは機能喪失変異(Loss of Function variant = LoF)と呼ばれます。
(注6)転写/翻訳/セントラルドグマ:体内でタンパク質が作られる際には、遺伝子に対応する部分のDNA配列情報からmRNAが作られ、そのmRNAの配列情報をもとにタンパク質が合成されます。mRNAが作られる前者の過程は「転写」、タンパク質が合成される後者の過程は「翻訳」と呼ばれ、DNA配列→mRNA→タンパク質の順に遺伝情報が伝達されタンパク質が作られる過程はセントラルドグマと呼ばれます。
(注7)遺伝的背景:英語におけるgenetic background、すなわちゲノム情報全体から定義される個人や集団の特徴付けのこと。
問い合わせ先
〈コロナ制圧タスクフォースに関するお問い合わせ先〉
慶應義塾大学医学部
教授 (内科学教室(呼吸器))福永 興壱(ふくなが こういち)
https://www.keio-pulmonary.jp/staff/fukunaga/
専任講師 (感染症学教室) 南宮 湖(なむぐん ほう)
https://infection.med.keio.ac.jp/staff/151/
慶應義塾大学医学部
教授 (内科学教室(呼吸器))福永 興壱(ふくなが こういち)
https://www.keio-pulmonary.jp/staff/fukunaga/
専任講師 (感染症学教室) 南宮 湖(なむぐん ほう)
https://infection.med.keio.ac.jp/staff/151/
〈本研究の内容に関するお問い合わせ先〉
東京大学大学院医学系研究科 遺伝情報学
教授 岡田 随象(おかだ ゆきのり)(大阪大学大学院医学系研究科 兼任)
https://www.u-tokyo.ac.jp/focus/ja/people/k0001_05137.html
准教授(研究当時)王 青波(おう せいは)
〈JST事業に関するお問い合わせ先〉
科学技術振興機構 戦略研究推進部 ライフイノベーショングループ
https://www.jst.go.jp/
慶應義塾大学信濃町キャンパス総務課
https://www.keio.ac.jp/ja/
大阪大学 大学院医学系研究科 広報室
https://www.med.osaka-u.ac.jp/
東京大学大学院医学系研究科 総務チーム
https://www.m.u-tokyo.ac.jp/index.html
理化学研究所 広報室 報道担当
https://www.riken.jp/
科学技術振興機構 広報課
https://www.jst.go.jp/
