| 発表のポイント |
|---|
|
発表概要
東京大学医科学研究所機能解析イン・シリコ分野の中井謙太教授とマルティン・ローザ助教は、京都大学医生物学研究所組織恒常性システム分野のアレクシス・バンデンボン准教授との共同研究で、ヒトゲノム上のハウスキーピング制御配列(注1)と一部のがん抑制遺伝子との関連を明らかにしました。ヒトゲノムは約30億文字の塩基の並び(配列)で構成されていますが、その中で通常遺伝子といわれるタンパク質情報の記載領域はごくわずかで、残りの大部分の果たす役割に関心がもたれています。そこには、主に遺伝子の読み取り開始場所を指定するプロモーター配列と、細胞の種類に応じてそのプロモーター配列と相互作用して遺伝子のオンオフを制御するエンハンサー配列が含まれます。
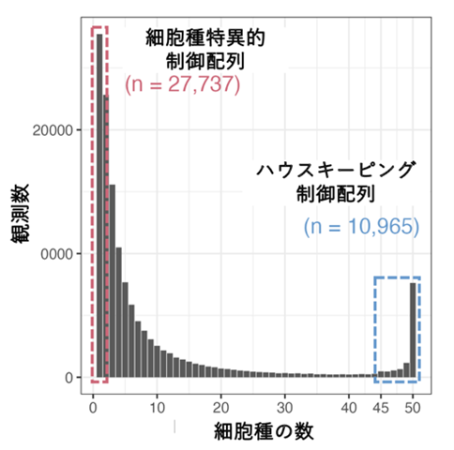
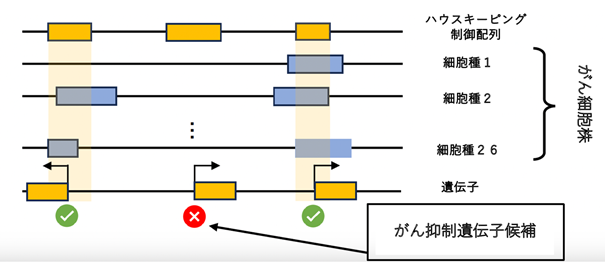
今回、エンハンサーがどの細胞で活性化しているかを網羅的に調べることで、約1万1千の領域がほとんど全ての細胞で活性化しているハウスキーピング制御配列であることを発見しました。それらは、エンハンサーというよりは、従来プロモーターとされている領域にありましたが、通常の意味でのプロモーターとは違って、周囲の多数の遺伝子の制御に関わる、むしろエンハンサー的な働きをしていることが示唆されました。さらに、それらの制御配列のいくつかは、がん細胞で高い確率で働いておらず、既知のがん抑制遺伝子の近くにあることがわかりました(図1)。
本研究により、ヒトゲノム上に存在するエンハンサー的プロモーター領域が網羅的に同定され、その機能の一端として、細胞のがん化を抑制する働きが明らかになりました。その結果、遺伝子の制御機構の理解が深まるとともに、この制御領域の状態をコントロールする新たながんの診断薬や治療薬の開発につながる可能性があります。
本研究成果は12月12日、国際科学雑誌「Nucleic Acids Research」(オンライン版)に公開されました。
発表内容
〈研究の背景〉ヒトゲノムが4種類の塩基のおよそ30億個の並び(配列)で構成されていることはよく知られていますが、通常、遺伝子といわれるタンパク質の構造情報を記載している領域は、そのうちの1.5%程度と言われており、残りの大部分の領域に書かれた情報の内容に興味がもたれています。そこには生体を構成する様々な細胞の種類に応じて、適切な遺伝子のオンオフを制御するシス制御領域と呼ばれる領域が含まれています。
シス制御領域は、さらに遺伝子情報記載の開始場所の情報を含むプロモーターと呼ばれる領域と、細胞の種類に応じて活性化状態を変化させ、それによって周囲のプロモーター領域との相互作用を調節することで、遺伝子のオンオフの切り替えに関わるエンハンサーと呼ばれる領域を含んでいます。
通常、プロモーターはその近傍の遺伝子一個の制御に関わるのに対して、エンハンサーは遺伝子よりもずっと多数存在します。さらに複数のエンハンサーが一つの遺伝子の制御に関わったり、逆に一つのエンハンサーが複数の遺伝子の制御に関わったりしますが、その詳細はよくわかっていません。エンハンサーは細胞の種類を特徴づけるために重要で、これまで個々の細胞に特徴的に活性化されるエンハンサーが比較的よく研究されてきたものの、その全体像についてはよく知られていませんでした。一方、遺伝子の中には、すべての細胞の生存に必須で、どの細胞でもオンになっているハウスキーピング遺伝子があり、ヒトではおよそ2,000個余りが知られています。しかしそれらの制御機構についてはあまり研究されていませんでした。
〈研究の内容〉
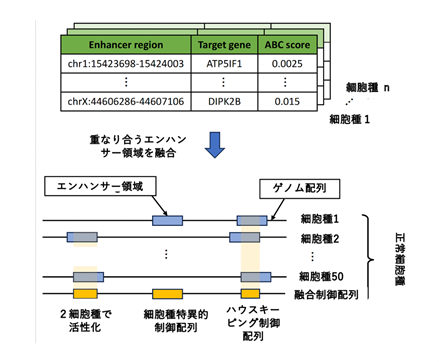
一般に、それぞれの種類の細胞でどのエンハンサーがどの遺伝子を制御しているかを網羅的に知ることは容易ではありません。しかしゲノムDNAやその結合タンパク質の化学修飾状態などのエピゲノム情報から推定することはある程度可能で、100種類以上の細胞についての予測データが公開されています。
今回、本研究グループは、その中のまず50種類の正常細胞のデータを比較して、ゲノム上のそれぞれのエンハンサーが何種類の細胞で活性化されているかを検討しました(図2)。その結果、大多数のエンハンサーは、1種類の細胞でしか活性化されていないのに対して、およそ1万1千個のエンハンサーはほとんどすべての細胞で活性化されていることがわかりました。よく調べてみると、それらのエンハンサーは、これまでプロモーター領域とされている領域と重なっていて、エピゲノム情報も通常のエンハンサーとは異なるため、これらをハウスキーピング制御配列と呼ぶことにしました。しかし、このハウスキーピング制御配列の存在位置は、ゲノム上でタンパク質コード遺伝子が密集している位置とよく相関していることや、上述の予測結果などから、これらの制御配列は通常のプロモーター配列とは異なり、周囲の多数の遺伝子(のプロモーター)と相互作用していることが示唆されました。
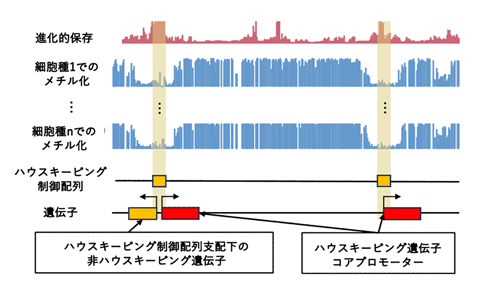
さらに、最初にエンハンサー領域の特徴づけに使った50種類の細胞データ以外に含まれている正常細胞のデータとがん細胞のデータについて、ハウスキーピング制御配列の活性化状態を調べてみました。すると、正常細胞については、予想通り、ほとんどのハウスキーピング制御配列が活性化されていたのに対して、がん細胞では、一部のハウスキーピング制御配列の活性化状態が大きく損なわれており、周囲の遺伝子の読み取りも不活化されていることがわかりました(上図1)。興味深いことに、それら、がん細胞で多く機能低下を示すハウスキーピング制御配列は、第19番染色体のサブテロメア領域(注2)に顕著に観察されました。さらに、その近傍にはがん抑制遺伝子として報告されているジンクフィンガー型転写因子(注3)などが特徴的に存在することがわかりました。
〈今後の展望〉
ゲノム上に多数存在する遺伝子スイッチの制御領域については、まだまだわからないことが多く、今回の研究により、新たな切り口が明らかになったと考えられます。また、今回の研究が、生の細胞やDNAを用いず、多くの公共データを組み合わせた、純粋な情報学的アプローチから得られた成果であることも特筆されます。今回の結果は、がん細胞で一部のハウスキーピング制御配列が不活性化されることで、周囲のがん抑制遺伝子の働きが抑えられることが発がんやがんの進行に関わっているとすれば、うまく説明できます。もし、この仮説が正しければ、このメカニズムをもとに新たながんの治療法や診断法、創薬などにつながる可能性もあり、実験的検証など、今後の研究の展開が期待されます。
発表者
東京大学医科学研究所 附属ヒトゲノム解析センター 機能解析イン・シリコ分野
中井 謙太(教授)
マルティン・ローザ(助教)
京都大学医生物学研究所 組織恒常性システム分野
アレクシス・バンデンボン(准教授)
論文情報
〈雑誌〉 Nucleic Acids Research
〈題名〉 Epigenetic characterization of housekeeping core promoters and their importance in tumor suppression
〈著者〉 Martin Loza, Alexis Vandenbon, and Kenta Nakai*
*責任著者
〈DOI〉 10.1093/nar/gkad1164
研究助成
本研究は、科研費「基盤研究(C)(課題番号:22K06189)」、「研究活動スタート支援(課題番号:22K21301)」の支援により実施されました。
用語解説
(注1)ハウスキーピング制御配列今回の研究で報告された、体内のほとんどすべての細胞において活性化状態にあり、エンハンサーとプロモーターの性質を併せ持つような、ゲノム上の遺伝子制御配列。
(注2)サブテロメア領域
ヒト染色体両端には特徴的な繰り返し配列からなるテロメアと呼ばれる領域があるが、テロメアと通常の染色体DNAの間には、サブテロメアという、これもまた繰り返し配列に富んだ領域があり、そこには多くの遺伝子ファミリーが存在する。
(注3)ジンクフィンガー型転写因子(遺伝子スイッチのオンオフを制御するタンパク質)
転写因子とは、特定のDNA塩基配列を認識して結合し、遺伝子スイッチのオンオフを制御するタンパク質のこと。いくつかのグループに分類されるが、その一つにジンクフィンガーという亜鉛イオンを含む特徴的な構造をもつグループがある。
問合せ先
〈研究に関する問合せ〉
東京大学医科学研究所 附属ヒトゲノム解析センター 機能解析イン・シリコ分野
教授 中井 謙太(なかい けんた)
〈報道に関する問合せ〉
東京大学医科学研究所 プロジェクトコーディネーター室(広報)