| 発表のポイント |
|---|
|
発表概要
東京大学医科学研究所附属ヒトゲノム解析センター 健康医療インテリジェンス分野の井元清哉教授・佐藤憲明助教の研究グループは、大阪公立大学大学院医学研究科 ゲノム免疫学の植松智教授(東京大学医科学研究所附属ヒトゲノム解析センター メタゲノム医学分野 特任教授を兼務)らとの共同研究を行い、Kyoto Encyclopedia of Genes and Genomes (KEGG)に蓄積された情報を用いてマルチオミクスデータ(注1)の可視化・解析を行う基盤的なRパッケージ ggkegg(注2) を開発しました。KEGGは、システム生物学の理解を容易にするためにデザインされた包括的なツールとデータベースを提供しており、遺伝子やタンパク質の機能、代謝経路、シグナル伝達、疾患の分子基盤の解析など、多様な研究分野で広く利用されています。本研究成果は10月16日(日本時間)にバイオインフォマティクス分野の国際科学雑誌『Bioinformatics』にオンライン掲載されました。発表内容
〈研究の背景〉現在、KEGGデータベースには、25,000を超えるオーソログ(注3)情報と、複数のオーソログ間の関係を機能別に整理した500を超えるパスウェイが存在しており、このような膨大かつ複雑な生物学的知識をデータ解析に活用するには、ネットワークベースの解析と解析目的に応じたカスタマイズ可能な結果の可視化が必要です。
これまでにも、KEGGの公式ツールを含め、様々なデータの可視化・解析パッケージが開発されてきましたが、パスウェイ以外のKEGG情報の活用や、代謝経路全体を俯瞰したパスウェイのカスタマイズされた可視化、他のパッケージから出力された解析結果とKEGG情報の統合といった機能は完全にはサポートされていませんでした。
〈研究の内容〉
本研究では、よりフレキシブルな可視化や、他のオミクス解析パッケージとの連携、ネットワークベースの解析を促進する特徴を持つRパッケージ ggkegg を開発しました。ggkeggはgrammar of graphicsによる可視化の統合的なライブラリであるggplot2を基にKEGGデータを解析・可視化するパッケージです。
Grammar of graphicsとは、グラフィックの構成要素を簡潔に記述するものであり、KEGGに含まれる遺伝子や酵素といった情報を1つ1つレイヤーとして構成することで複雑なKEGGデータの解析を容易に行うことができます。公共データベースに登録されているバルク・シングルセルトランスクリプトームデータ(注4)の解析と、メタゲノムデータ解析においてこのパッケージを利用した結果、このパッケージがオミクスデータのより深い理解を助け、ユーザーのニーズに対応した情報の可視化を行えることを示しました。
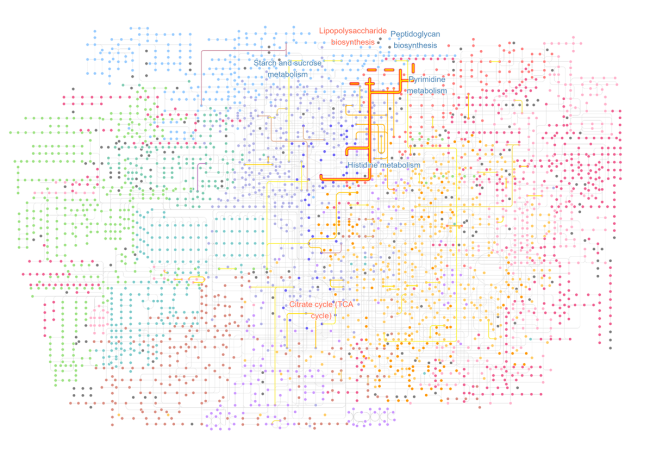
例として、トランスクリプトーム解析において複数のデータセットから得られた統計情報を基に、複数のKEGGパスウェイ情報とネットワーク解析を用いて重要な遺伝子クラスターを同定できることを示しました。また、公共データベースに登録されているクローン病患者の腸内細菌叢メタゲノムデータから、患者と健常者で差異のある代謝経路をKEGGの包括的な代謝経路情報に反映し、研究結果の効果的な可視化が可能なことを示しました(図)。
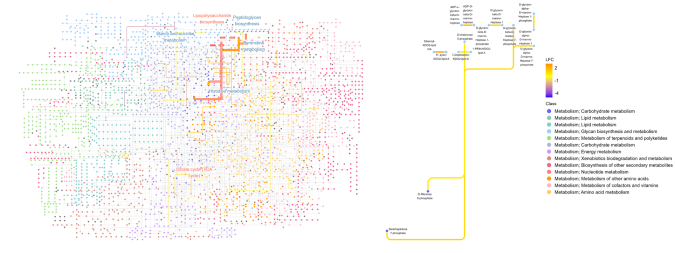
公共データベースに登録されているクローン病患者の腸内細菌叢メタゲノムデータから、患者と健常者で差異のある代謝経路をKEGGの代謝経路上に反映した(左)。その内、興味のあるリポポリサッカライドの生合成に関連した経路の詳細を化合物名と共に拡大して表示した(右)。俯瞰的、個別に代謝経路を解析することでオミクスデータの理解を促進する。
様々なオミクス解析に我々の開発したggkeggを用いることで、膨大なKEGG情報を効率的に活用しながら得られたオミクスデータの生物学的な解釈をより深めることができると考えられます。パッケージはオープンソースで公開されており(https://github.com/noriakis/ggkegg)、生物学的データ解析パッケージを収載したリポジトリであるBioconductorからインストール可能で簡便に利用することが可能です。幅広い研究者に利用されることで生命科学研究開発の促進に繋がることが期待されます。
発表者
東京大学医科学研究所附属ヒトゲノム解析センター 健康医療インテリジェンス分野
井元 清哉(教授)
佐藤 憲明(助教)
大阪公立大学大学院医学研究科 ゲノム免疫学
植松 智(教授)〈東京大学医科学研究所附属ヒトゲノム解析センター メタゲノム医学分野 特任教授を兼務〉
論文情報
〈雑誌〉 Bioinformatics
〈題名〉 ggkegg: analysis and visualization of KEGG data utilizing the grammar of graphics
〈著者〉 Noriaki Sato, Miho Uematsu, Kosuke Fujimoto, Satoshi Uematsu, Seiya Imoto*
*責任著者
*責任著者
〈DOI〉 10.1093/bioinformatics/btad622
用語解説
生体中に存在する分子全体の網羅的なデータ。メタゲノミクスやトランスクリプトミクスなど、複数のオミクスデータを指す。
(注2)Rパッケージ ggkegg
今回開発したパッケージで、統計計算とグラフィックスのためのフリーソフトウェア環境であるR内で利用できる。
(注3)オーソログ:
異なる種で共通の祖先遺伝子から生じた相同な遺伝子。
(注4)バルク・シングルセルトランスクリプトーム
バルクトランスクリプトームはサンプル中の全細胞のメッセンジャーRNA発現を測定し、シングルセルトランスクリプトームは個々の細胞における発現を測定する。
問合せ先
〈研究に関する問合せ〉
東京大学医科学研究所附属ヒトゲノム解析センター
教授 井元 清哉(いもと せいや)
https://www.ims.u-tokyo.ac.jp/imsut/jp/lab/hgclink/page_00072.html
〈報道に関する問合せ〉
東京大学医科学研究所 プロジェクトコーディネーター室(広報)
https://www.ims.u-tokyo.ac.jp/
東京大学医科学研究所附属ヒトゲノム解析センター
教授 井元 清哉(いもと せいや)
https://www.ims.u-tokyo.ac.jp/imsut/jp/lab/hgclink/page_00072.html
〈報道に関する問合せ〉
東京大学医科学研究所 プロジェクトコーディネーター室(広報)
https://www.ims.u-tokyo.ac.jp/