本研究成果は、アトピー性皮膚炎の治療標的となる病態の解明につながると期待できます。
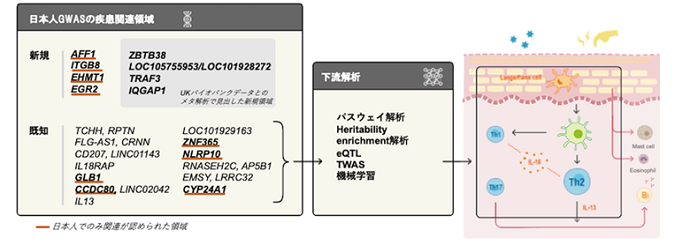
アトピー性皮膚炎は世界的に発症頻度の高いアレルギー性疾患であり、その高い遺伝率から、遺伝的要因が病態形成に大きく影響すると考えられています。今回、研究グループは、バイオバンク・ジャパン(BBJ)[2]に登録された約12万人のデータを対象に、アジア人のアトピー性皮膚炎としては最大規模のGWASを行い、四つの新しい疾患関連領域を同定しました。加えて、UKバイオバンク[3]のGWAS結果とのメタ解析[4]により、さらに四つの新たな疾患関連領域を同定しました。また、NLRP10領域とCCDC80領域については、数多くの多型の中から原因多型の可能性の高いものを同定しました。同定した二つの一塩基多型(SNP)[5]は日本人でのみ疾患との関連が認められています。その一方で、日本人とヨーロッパ人に共通して、免疫細胞や皮膚細胞がアトピー性皮膚炎の遺伝的要因に関わる主要な細胞種であることが明らかになりました。
本研究は、科学雑誌『Journal of Allergy and Clinical Immunology』(6月8日付)に掲載されました。
背景
アトピー性皮膚炎は世界的に発症頻度の高いアレルギー性疾患であり、その高い遺伝率から注1)、遺伝的要因がその病態形成に大きく影響すると考えられています。近年、他の多くの疾患と同様にアトピー性皮膚炎に対しても、ゲノムワイド関連解析(GWAS)などの遺伝学的解析が行われており、アトピー性皮膚炎の疾患関連領域が多く同定されていますが、アトピー性皮膚炎の遺伝学的背景を説明する材料としては不十分でした。特に、アジア人の解析で同定された疾患関連領域は少なく、その中でも特に病態に関わる一塩基多型(SNP)は特定されていませんでした。
そこで、研究グループは日本人を対象に大規模なGWASを行い、さらにUKバイオバンクのGWAS結果を用いてメタ解析を行うことで、新たな疾患関連領域を探索しました。
注1)Elmose C, Thomsen SF. Twin Studies of Atopic Dermatitis: Interpretations and Applications in the Filaggrin Era. J Allergy (Cairo) 2015; 2015:902359.
研究手法と成果
研究グループは、バイオバンク・ジャパン(BBJ)の登録者のうち、アトピー性皮膚炎患者2,639人とコントロール群11万5648人(計11万8287人)を対象にGWASを行いました。その際、DNAマイクロアレイ[6]のデータからさらに多くの遺伝的バリアント(個人間のゲノムDNA配列の違いがある部分)の情報を推定するインピュテーション法[7]の精度を上げるため、インピュテーション法に用いる参考配列を約3,000人分の日本人特有の低頻度、あるいは稀な遺伝的バリアントを多く含むデータにしました。これにより、従来よりも多くの遺伝的バリアントを解析対象とすることができました。
解析の結果、過去に行われたBBJのアトピー性皮膚炎のGWAS注2)よりも9個多い、17個の疾患関連領域を同定しました。そのうち、4領域(AFF1、ITGB8、EHMT1、EGR2)はこれまで報告されていないものでした(図1)。今回新たに同定した疾患関連領域中のSNPは、ヨーロッパ人では極めて頻度が低く、日本人においても頻度が低かったことから、日本人の大規模解析を行ったからこそ見つけられた関連領域であると考えられます。ほかにも、これまでヨーロッパ人の解析では報告されたものの、アジア人では不明だったIL13などの領域にも関連を見いだし、ヨーロッパ人とアジア人で疾患発症に関わる共通の遺伝的背景を明らかにしました。また、遺伝子単位で遺伝的バリアントの情報を統合したうえで解析したところ、免疫に関わる重要な転写因子[8]SMAD4も疾患に関連することが分かりました。
注2)Hirota T, Takahashi A, Kubo M, Tsunoda T, Tomita K, Sakashita M, et al. Genome-wide association study identifies eight new susceptibility loci for atopic dermatitis in the Japanese population. Nat Genet 2012; 44:1222-6.
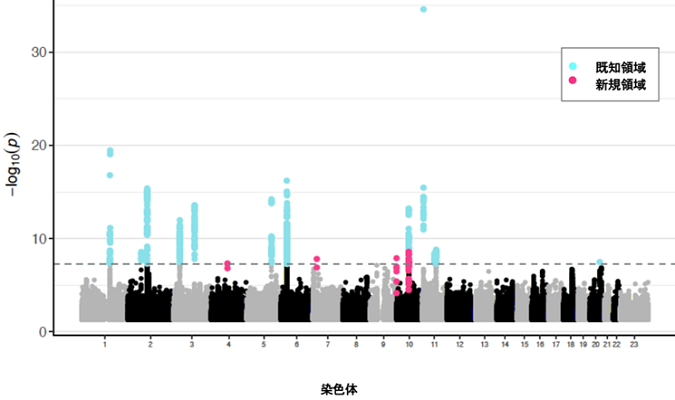
解析対象となった全ゲノム領域のp値をプロット。点線がp 値=5.0×10-8のゲノムワイド有意水準に該当する。今回、全部で17領域が有意水準を満たし、そのうちピンクで示す四つが新規領域であった。
また、UKバイオバンクのデータとともにメタ解析を行った結果、さらに新たな疾患関連領域として4領域(ZBTB38、LOC105755953/LOC101928272、TRAF3、IQGAP1)を同定しました(図2)。これらは、ヨーロッパ人集団と共通した疾患関連領域であるために見つかったと考えられます。
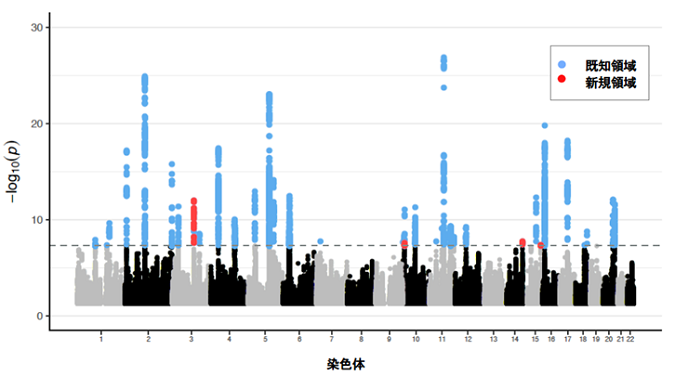
解析対象となった全ゲノム領域のp値をプロット。点線がp値=5.0×10-8のゲノムワイド有意水準に該当する。今回、全部で39領域が有意水準を満たし、そのうち赤で示す四つが新規領域であった。
次に、これらの疾患関連領域のうちSNPが影響を及ぼす疾患関連メカニズムを調べたところ、自然免疫に関連するNLRP10領域にアミノ酸配列に変化を起こすミスセンス変異[9]がありました。このミスセンス変異が存在するNACHTドメインは、過去に免疫反応に重要な転写因子TNIP1の結合部位として報告されており、このドメイン内の変異がアトピー性皮膚炎における免疫反応にも影響を持つ可能性が示されました。
また、理研の研究チームが2020年に開発した機械学習[10]の手法を用いて注3)、遺伝制御領域[11]の活性に影響を与えるSNPを調べました。この手法では、従来の遺伝子発現とSNPの関連解析(eQTL)[12]では検索しきれない稀な多型について、FANTOM5注4)プロジェクトで同定された遺伝子制御領域の活性への直接的な効果を細胞種ごとに推測できます。今回、日本人のアトピー性皮膚炎との関連があったSNPに、CCDC80領域のエンハンサー[11]に重なるものが複数存在しましたが、機械学習による解析からそのエンハンサー活性を変化させるSNP(rs12637953)があることが分かり、その影響は皮膚細胞で認められました(図3A, B)。CCDC80は皮膚や脂肪組織内の酸化反応に関わるタンパク質で、マウスのアトピー性皮膚炎のモデルにおいて、アレルギー反応の発生に関わることが報告されています注5)。また実際に、実験でこのSNPが近隣のプロモーター[11]の発現を変化させることを示しました(図3C)。このように、rs12637953が遺伝子制御領域の発現調整を介することで、疾患と関わることが示唆されました。
NLRP10領域とCCDC80領域は、過去にも日本人のGWASでのみ疾患との関連が認められた領域です。また、今回同定したこの二つのSNPに関しては、日本人とヨーロッパ人との間に頻度差があり、日本人では頻度が高いのに対して、ヨーロッパ人では非常に稀であることが分かりました。これらのことから、この二つのSNPは日本人のアトピー性皮膚炎の発症に関わる可能性が高いと考えられます。
注3)Koido M, Hon C-C, Koyama S, Kawaji H, Murakawa Y, Ishigaki K, et al. Predicting cell- type-specific non-coding RNA transcription from genome sequence. bioRxiv 2020:2020.03.29.011205.
注4)2014年3月27日プレスリリース「ゲノム上の遺伝子制御部位の活性を測定し正常細胞の状態を定義」
https://www.riken.jp/press/2014/20140327_1/index.html
注5)Jung K, Tanaka A, Fujita H, Matsuda A, Oida K, Karasawa K, et al. Peroxisome proliferator-activated receptor γ-mediated suppression of dendritic cell function prevents the onset of atopic dermatitis in NC/Tnd mice. J Allergy Clin Immunol 2011; 127:420-9.e1-6.
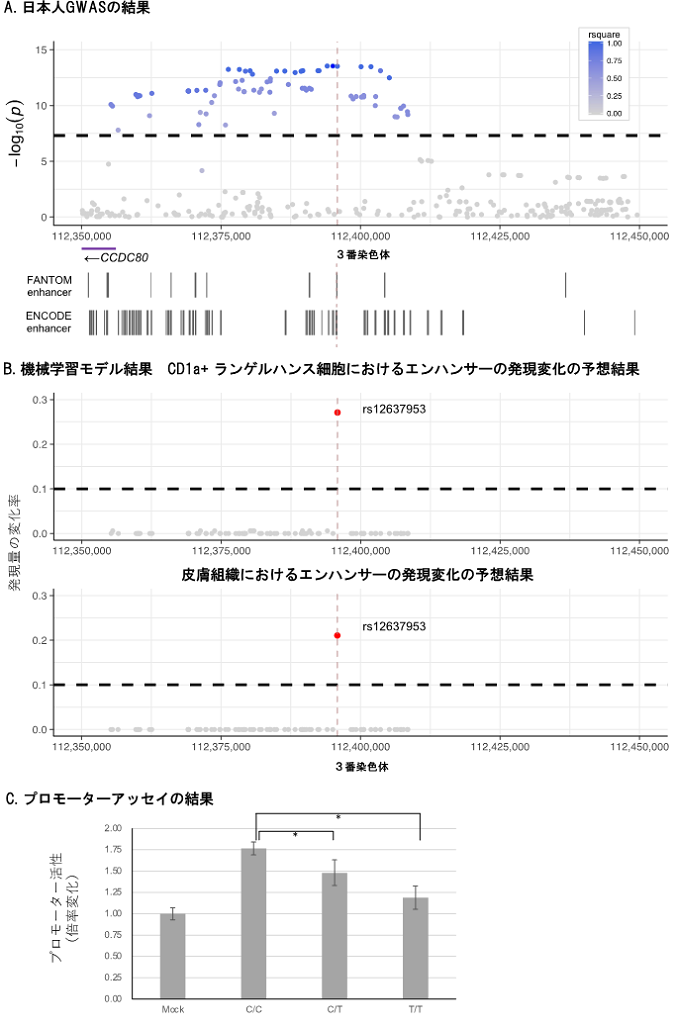
A: 日本人のアトピー性皮膚炎のGWASで、CCDC80の遺伝子間領域に有意領域を認めた。rs12637953は、FANTOMプロジェクトとENCODEプロジェクトの両方でエンハンサー上に存在するSNPであることが報告されている。
B: Aのプロットに含まれるSNPが機械学習モデルでプローターやエンハンサーの発現に影響するかどうかを検索した。結果、rs12637953がエンハンサー発現に強い影響を持つことが予想された。
C: rs12637953の多型がプロモーターアッセイで近隣のプロモーター活性に影響を与えるかの実験。Referenceアレル(C)に比較して、Alternativeアレル(T)がプロモーター活性を低下させることが分かった。
上記のように、一つのSNPレベルで疾患に関わるものを特定した一方で、全てのSNPの影響を総合的に評価すると、日本人とヨーロッパ人に共通して、アトピー性皮膚炎に関連する遺伝的多型が免疫細胞のCD4 T細胞[13]の遺伝制御領域に集積していることが分かりました(図4)。また、両人種のGWASから見つかった疾患関連領域が、CD4 T細胞および皮膚のケラチノサイト(角化細胞)[14]のエンハンサー領域と重なることが分かりました。さらにパスウェイ解析[15]においても、CD4 T細胞関連のパスウェイと関連があることが分かりました。CD4 T細胞やケラチノサイトは、アトピー性皮膚炎の病態にとって主要な細胞と考えられてきましたが、遺伝要因の関与は明確には分かっていませんでした。この結果により、CD4 T細胞やケラチノサイトでの遺伝子発現が、アトピー性皮膚炎の発症に関わることが示唆されました。
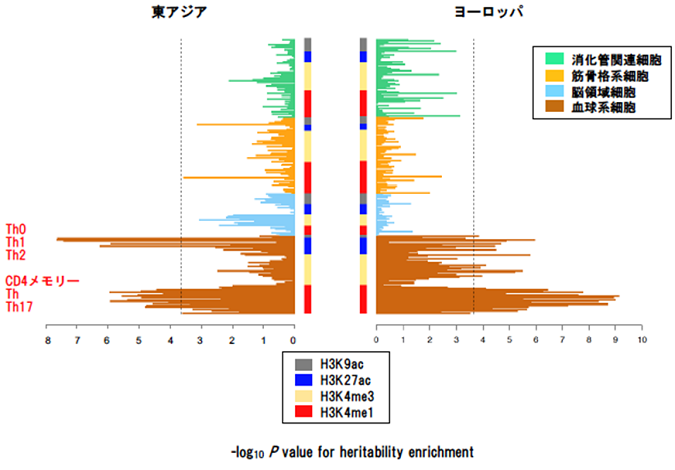
日本人、UKバイオバンクのそれぞれのGWAS結果をもとに、アトピー性皮膚炎の疾患関連変異がどの細胞の遺伝子発現制御領域に集積しているかを解析した。両方の人種で、血球細胞に高い集積が見られ、その中でも、CD4 T細胞のサブセットのH3K4me1/H3K27acに強い集積が見られた。H3K4me1/H3K27ac領域は、エンハンサー領域のマーカーと考えられている。
また、多型によって規定される細胞ごとの遺伝子発現を全ゲノム領域において推定し、それらがアトピー性皮膚炎の発症と関連があるかどうかを調べるトランスクリプトームワイド関連解析(TWAS)[16]を行いました。その結果、複数の血液細胞(CD4、NK、好中球)におけるIL18受容体(IL18R)の発現量の違いが疾患に関わることが分かりました。IL18受容体の種類や効果量は、細胞によって異なりました。IL18は慢性炎症期や自然免疫において重要な役割を担うことが知られており、アトピー性皮膚炎にも影響すると推測されていました。IL18R領域はこれまでのGWASでも関連があることが知られていましたが、その詳細は分かっていませんでした。
今回、複数の遺伝的要因によってIL18Rの発現量が変わり、その違いを介してアトピー性皮膚炎に関わることが示唆され、細胞種によってさまざまなパターンをとることが分かりました。具体的には、CD4 T細胞におけるIL18R1の減少、NK細胞におけるIL18R1の増加、好中球におけるIL18RAPの減少がアトピー性皮膚炎の発症リスクにつながることが分かりました。
以上のように、日本人とヨーロッパ人に共通して、Th1、Th2、Th17といったCD4 T細胞やケラチノサイトに関連する遺伝的要因がアトピー性皮膚炎の発症に関わっていること、また日本人においては、NLRP10、CCDC80、IL18受容体が重要であることが分かりました。
今後の期待
今回の研究では、インピュテーション法などのGWASの手法を改善することで、日本人における新たなアトピー性皮膚炎の疾患関連領域を同定しました。また、単に疾患関連領域を同定するだけでなく、遺伝的に疾患に関連する細胞やタンパク質も同定しました。今後、さらにアトピー性皮膚炎の遺伝学的要因を調べていくことで、治療標的となりうる病態が解明されると期待できます。論文情報
<タイトル>Eight novel susceptibility loci and putative causal variants in atopic dermatitis
<著者名>
Nao Tanaka, Masaru Koido, Akari Suzuki, Nao Otomo, Hiroyuki Suetsugu, Yuta Kochi, Kouhei Tomizuka, Yukihide Momozawa, Yoichiro Kamatani, The Biobank Japan Project, Shiro Ikegawa, Kazuhiko Yamamoto, and Chikashi Terao.
<雑誌>
Journal of Allergy and Clinical Immunology
補足説明
[1] ゲノムワイド関連解析(GWAS)形質に対する遺伝的関連を知るための手法であり、SNP(一塩基多型)を用いて解析するものが一般的である。形質(疾患のある/なしや量的形質)を目的変数、SNPの量的情報や各種共変量を説明変数にしてモデル化し、SNPの関連を評価する。GWASはGenome-Wide Association Studyの略。
[2] バイオバンク・ジャパン(BBJ)
日本人集団27万人を対象とした生体試料のバイオバンクで、東京大学医科学研究所内に設置されている。理化学研究所が実験を行って取得した約20万人のゲノムデータを保有する。オーダーメイド医療の実現プログラムを通じて実施され、ゲノムDNAや血清サンプルを臨床情報とともに収集し、研究者へのデータ提供や分譲を行っている。
[3] UKバイオバンク(UKB)
英国で構築されているバイオバンクであり、50万人規模の疾患罹患情報、臨床情報、遺伝情報などから構成される。
[4] メタ解析
二つ以上の統計解析の結果を合わせる際に、それぞれの解析結果でばらつきがある面を統計学的に排除し、偏りのない合算をする手法。
[5] 一塩基多型(SNP)
一つの遺伝的座位に、二つかそれ以上の頻度の高い異なるアレルが存在する状態のことを遺伝的多型という。一つの塩基がほかの塩基に変わる多型を、一塩基多型と呼ぶ。SNPはSingle Nucleotide Polymorphismの略。
[6] DNAマイクロアレイ
基板の上に、遺伝的多型(主にSNP)に相補的なprobeを搭載したビーズを高密度に配置し、数十万~数百万の遺伝的多型を検出するための分析器具。
[7] インピュテーション法
DNAマイクロアレイで一部の遺伝多型を測定した後に、そこで得られた遺伝型を用いて実験的には測定していない遺伝的変異を推定し、補完する遺伝統計学的手法。参照配列として、1000ゲノムプロジェクトで解明された配列が世界的に用いられているが、今回は日本人の全ゲノムシークエンスデータの参照配列を用いた。
[8] 転写因子
DNAに配列特異的に結合するタンパク質で、転写制御領域に結合し、RNAポリメラーゼによる遺伝子の転写を活性化あるいは不活性化する。
[9] ミスセンス変異
ゲノムの塩基配列によりアミノ酸のコードが変化し、本来のアミノ酸ではないアミノ酸で置き換わったタンパク質が生成される変異。
[10] 機械学習
データをコンピュータに入力し、その中にある規則性を発見させることで、未知のデータに対する解答を得る手法。
[11] 遺伝制御領域、エンハンサー、プロモーター
ゲノム配列上の、遺伝子の発現を制御する機能を持つ領域。エンハンサーやプロモーターなどの種類が知られている。プロモーターは遺伝子の転写開始点の近くで遺伝子発現を制御する領域であり、エンハンサーはプロモーターより離れた領域でプロモーターや遺伝子領域の発現を制御する領域である。
[12] 遺伝子発現とSNPの関連解析(eQTL)
SNPと遺伝子発現量の関連解析によって、遺伝子発現に関わるゲノム領域を特定する。eQTLはexpression Quantitative Trait Locusの略。
[13] CD4 T細胞
リンパ球の一種。CD4陽性T細胞は、他の種類のリンパ球の分化成熟を調整し、免疫反応システムの司令塔としての役割を持つ。
[14] ケラチノサイト(角化細胞)
皮膚表皮を形成する上皮細胞。最も内側の基底層に存在する角化幹細胞が存在は、分化しながら外側の層へと徐々に位置を移し、表皮組織に必要なさまざまな物質を産生する。
[15] パスウェイ解析
シグナル伝達系、遺伝子の制御関係、代謝経路、タンパク質間相互作用などの情報をもとにグループ化された遺伝子やタンパク質の集合のことをパスウェイと呼ぶ。過去に分類されたパスウェイごとの遺伝子が疾患に関連があるかどうかを解析する手法のことをパスウェイ解析と呼ぶ。
[16] トランスクリプトームワイド関連解析(TWAS)
多型によって規定される遺伝子発現を全ゲノム領域において予想し、それらが、疾患の発症と関連があるかどうかを解析する手法。TWASはTranscriptome-Wide Association Studyの略。
発表者・機関窓口
<発表者>※研究内容については発表者にお問い合わせください。理化学研究所 生命医科学研究センター
https://www.ims.riken.jp/
ゲノム解析応用研究チーム
チームリーダー 寺尾 知可史(てらお ちかし)
(静岡県立総合病院 免疫研究部長、静岡県立大学 特任教授)
大学院生リサーチ・アソシエイト 田中 奈緒 (たなか なお)
自己免疫疾患研究チーム
チームリーダー 山本 一彦 (やまもと かずひこ)
<機関窓口>
理化学研究所 広報室 報道担当
https://www.riken.jp/
東京大学 大学院新領域創成科学研究科 広報室
https://www.k.u-tokyo.ac.jp/
静岡県立総合病院 総務課
http://www.shizuoka-pho.jp/sogo/
静岡県立大学 広報・企画室
https://www.u-shizuoka-ken.ac.jp/
日本医療研究開発機構 ゲノム・データ基盤事業部 ゲノム医療基盤研究開発課
