| 発表のポイント |
|---|
|
発表概要
東京大学医科学研究所 附属ヒトゲノム解析センター 健康医療インテリジェンス分野の井元 清哉教授、メタゲノム医学分野の植松 智 特任教授、株式会社DeNAライフサイエンス(以下、DeNAライフサイエンス)、森永乳業株式会社らの研究グループは、DeNAライフサイエンスが推進するユーザー参加型のゲノム研究プロジェクト「MYCODE Research」(マイコード・リサーチ)(注1)のもと、アジア人集団で初となる腸内細菌叢のゲノムワイド関連解析(GWAS、注2)を実施しました。腸内細菌叢は食事などの生活習慣(外的要因)や宿主遺伝要因(内的要因)の影響を受けることが知られていましたが、これまでは外的要因に着目した研究が主であり、内的要因も含めた詳細な解析は多くは報告されていませんでした。本研究では、MYCODE Researchによる研究参加者リクルートを行い、より均質な遺伝的背景を有する日本人集団に対して、外的要因の影響を調整したうえでGWASを実施したところ、関連が示唆される5つのゲノム領域を新規に同定しました。更に遺伝率(注3)の解析から、日本人腸内細菌叢の遺伝要因が多遺伝子構造(polygenic architecture、注4)を取ることを見いだしました。
本研究成果は、2020年11月18日に英国科学誌「Communications Biology」オンライン版に掲載されました。
発表内容
ヒトの腸管内には多種多様な細菌が生息しており、「腸内細菌叢(ちょうないさいきんそう)」と呼ばれる細菌群集がつくられています。腸内細菌叢の組成の違いが、様々な疾患の発症と関連することが明らかとなってきており、腸内細菌叢による疾患の早期診断や治療への応用が期待されています。一方で、健康な人の腸内細菌叢にも個人差が大きいことが知られています。腸内細菌叢は食事などの生活習慣(外的要因)や宿主遺伝要因(内的要因)の影響を受けることが知られていますが、これまでは外的要因に着目した研究が主であり、内的要因も含めた詳細な解析は多くは報告されていませんでした。また、遺伝要因の関与を調べる手法として、ゲノムワイド関連解析(GWAS)による網羅的な遺伝要因の探索が報告されていますが、これまで、欧米人集団を対象とした報告に限られていました。しかしながら、腸内細菌叢は民族など集団で異なることが知られていることから、欧米人集団に加えて、他の民族集団を対象とした遺伝要因の探索が求められていました。
日本人集団は、欧米人集団よりもゲノムの個人差が小さく、均質な遺伝的背景を持つと言われています。この特徴は、GWASによって腸内細菌叢とヒトゲノムとの関連をより正確に評価できる可能性があります。本研究では、より均質な遺伝的背景を有する日本人集団の特徴を活かし、アジア人集団で初となる、日本人腸内細菌叢のGWASによる遺伝要因の探索を試みました。
東京大学医科学研究所 附属ヒトゲノム解析センターとDeNAライフサイエンスは共同で、「健康長寿社会の実現」を目的として、DeNAライフサイエンスが提供する遺伝子検査サービス「MYCODE(マイコード)」を受けた会員のうち、研究に同意いただいている方を対象とした、ユーザー参加型のゲノム研究プロジェクト「MYCODE Research(マイコード・リサーチ)」を推進しています。
本研究では、MYCODE Researchのもと、インターネットを活用した研究参加者リクルートを行い、4 日間で2,000名近くの応募を集めました。その中から1,068人を対象に腸内細菌叢解析(注5)を行ない、取得済みの558,583のSNP(注6)データと併せてGWASを実施しました。
まず、被験者の外的要因である食習慣や生活習慣等138の項目についてのデータを取得し、それらと腸内細菌叢との関連性を評価しました。その結果、主要な21の菌について281の有意な関連性が認められました。これらの外的要因の影響を統計学的に調整したうえでGWASによる関連SNPの探索を精緻に行いました。
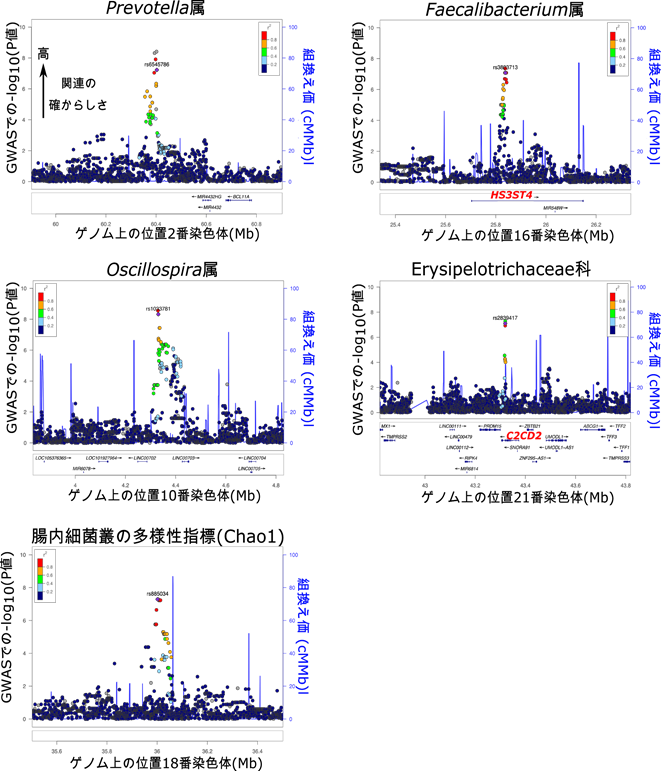
その結果、腸内細菌叢と関連する5つのゲノム領域を新規に同定することに成功しました(図1)。HS3ST4遺伝子領域のSNPは男性でのFaecalibacterium属という菌の割合と、C2CD2遺伝子領域上のSNPは女性でのErysipelotrichaceae科という菌の割合との関連が示されました。他3つのSNPは2番、10番、18番染色体上の遺伝子間領域に位置し、それぞれ男性でのPrevotella属という菌の割合、女性でのOscillospira属という菌の割合、女性における腸内細菌叢の多様性指標(注7)との関連を示しました。
各腸内細菌叢パラメーターに対するGWASの結果について、関連が示唆されるゲノム領域を示します。各点が解析したSNPを示します(直接調べたSNPと、imputation(インピュテーション)とよばれるコンピューター上で統計学的に推測したSNPを含みます)。左縦軸で示した-log10(P値)の値が大きいほど(P値が低いほど)関連が確からしいことを表します。各点の色分けで表示したr2値は、連鎖不平衡係数とよばれ、最も関連の確からしいSNP(紫色で表示しrs番号を並記)との相関を表します。この係数が大きいほど(暖色に近いほど)、そのSNPが、最も関連の確からしいSNPと同じシグナルを反映していることが示唆されます。灰色は連鎖不平衡係数が計算できなかったSNPです。青線は、右縦軸の組換え価と呼ばれる値を示します。この値が高いゲノム上の位置は、遺伝子組換えが起こりやすいことを表します。
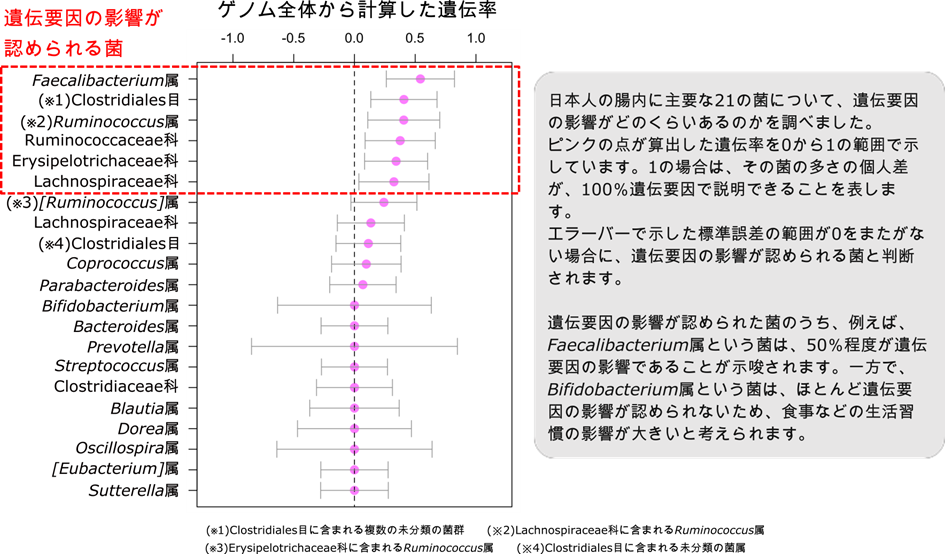
左に示すそれぞれの菌について、ゲノム全体から算出した遺伝率の推定値をピンクの点で表します。この値が大きいほど、遺伝要因の影響が大きいことを示唆し、赤破線で囲った6つの菌は、エラーバー(標準誤差)の範囲が0をまたがないことから、遺伝要因の影響が統計的に認められる菌であると判断されます。
また、我々が同定した日本人腸内細菌叢と関連を示すゲノム領域は、これまでに欧米人集団を対象としたGWASから報告されたゲノム領域とは異なる領域であったことから、人種特異的な遺伝要因の存在が示唆されました。
本研究で得られたGWASデータは、オープンデータとしてGWAS Catalog(注8)に登録を行いました。
本研究で得られた成果は、腸内細菌叢の民族差を理解する上で基礎的なデータとなることが期待されます。今後、宿主の遺伝的背景を考慮した上で、疾患や体質などの個々人の健康状態などと腸内細菌叢との関連性、更には民族性との繋がりに関してより精緻に評価する研究を検討していく予定です。
また、本研究の成果は、インターネットを活用することで、ユーザーコミュニティの個人が自らの同意の下で研究に参加して科学の発展に寄与できる、“Community-derived science”の実現により得られました。この新しい研究形態は、研究参加者リクルートやデータ収集に膨大な時間とコストがかかる従来型の研究の困難を解決し、研究成果の社会への還元を加速させることが期待されます。
発表雑誌
雑誌名:「Communications Biology」(オンライン版)論文タイトル:Genome-wide association studies and heritability analysis reveal the involvement of host genetics in the Japanese gut microbiota
著者:Sachiko Ishida*, Kumiko Kato, Masami Tanaka, Toshitaka Odamaki, Ryuichi Kubo, Eri Mitsuyama, Jin-zhong Xiao, Rui Yamaguchi, Satoshi Uematsu, Seiya Imoto, and Satoru Miyano
(*責任著者)
DOI:10.1038/s42003-020-01416-z
URL:https://www.nature.com/articles/s42003-020-01416-z
問い合わせ先
<研究に関するお問い合わせ>東京大学医科学研究所 健康医療インテリジェンス分野
教授 井元清哉(いもと せいや)
https://www.ims.u-tokyo.ac.jp/imsut/jp/lab/hgclink/page_00072.html
<報道に関するお問い合わせ>
東京大学医科学研究所 国際学術連携室(広報)
https://www.ims.u-tokyo.ac.jp/imsut/jp/
株式会社ディー・エヌ・エー 広報グループ 担当:山田
https://dena.com/jp/
森永乳業株式会社 広報IR部 担当:渡辺・見上
https://www.morinagamilk.co.jp/
用語解説
(注1)MYCODE Research(マイコード・リサーチ)株式会社DeNA ライフサイエンスの遺伝子検査サービス「MYCODE(マイコード)」会員の中から研究に同意いただいた方の協力を得て行うユーザー参加型の研究プロジェクト。↑
(注2)ゲノムワイド関連解析(GWAS:Genome Wide Association Study)
ヒトゲノム配列上に存在する数十~数百万か所のSNP等の遺伝子多型と疾患や体質との関連を網羅的に検討する、遺伝統計解析手法。数千人~百万人を対象に大規模に実施されることで、これまでに様々な疾患や体質に関連するがゲノム領域が同定されている。↑
(注3)遺伝率
疾患や体質といった形質に対する遺伝要因の影響度を測る尺度。形質の分散に対して遺伝要因の分散が占める割合で定義される。↑
(注4)多遺伝子構造(polygenic architecture)
疾患や体質といった形質に対して、寄与度はそれほど高くないSNP等の遺伝子多型が多数集積して影響を与える遺伝要因の様式。↑
(注5)腸内細菌叢解析
腸内細菌叢のDNA情報から含まれる菌やその割合を明らかにする解析手法。本研究では、細菌の16SリボソームRNA(rRNA)遺伝子を解析する方法(16S解析)を用いた。↑
(注6)SNP(single nucleotide polymorphism)
遺伝子多型と言われるDNA配列の個人差の一つ。ヒトが保有する約30億の塩基対の配列には人種や個人間で異なる部分があり、1つの塩基だけが別の塩基に置き換わっているものをSNPという。↑
(注7)多様性指標
腸内細菌の菌の種類の多さを評価する指標。多様性が極端に少ないと、炎症性腸疾患や肥満、糖尿病、アレルギーのリスクとなることが報告されている。↑
(注8)GWAS Catalog
さまざまな疾患、形質を対象としたGWASのデータがまとめられたカタログ。米国の国立ヒトゲノム研究所(National Human Genome Research Institute)と英国の欧州バイオインフォマティクス研究所(European Bioinformatics Institute)によって運営されている。↑
詳細は GWAS Catalogのホームページ(英語):https://www.ebi.ac.uk/gwas/